Entry Name alpha-galactosidase;
Class Hydrolases;BRITE hierarchy
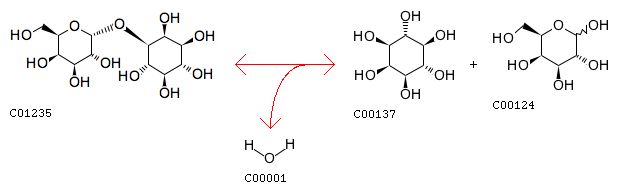
Sysname alpha-D-galactoside galactohydrolase
Reaction(IUBMB) Hydrolysis of terminal, non-reducing alpha-D-galactose residues in alpha-D-galactosides, including galactose oligosaccharides, galactomannans and galactolipids
Reaction(KEGG) Comment Also hydrolyses alpha-D-fucosides.
History EC 3.2.1.22 created 1961
Pathway ec00603 Glycosphingolipid biosynthesis - globo and isoglobo series
Orthology Genes MORG : 121436690 121437100
MFOT : 126493556 126503230
MDO : 100021325 100022779(GLA)
GAS : 123236359 123253328(GLA)
AAM : 106484973 106484974(GLA)
GSH : 117360984 117360986(GLA)
CCAR : 109053915 109087917
CAUA : 113075390 113081207 113114125
CGIB : 127971961 128027729
PPRL : 129360714 129360723
BPEC : 110158921 110159052
PSPA : 121295539 121326706
PVUL : 126830462 126830463
DPOL : 127875912 127876436
AQU : 100639143 100639269 100639555 100641521
ATH : AT3G56310 AT5G08370(AGAL2) AT5G08380(AGAL1)
ALY : 9307393 9309423 9312429
CRB : 17881987 17884048 17885543
CSAT : 104708252 104708254 104712293 104734858 104734859 104769045 104769047 104781530 104791927
EUS : EUTSA_v10005987mg EUTSA_v10013749mg EUTSA_v10013789mg
BRP : 103841539 103850693 103850695 103855779 103855780
BNA : 106367968 106368090 106380528 106380533 106384971 106384972 106396524 106396847 111204257 111210464
BOE : 106297579 106312208 106326722 106327837 106328866 106331447
RSZ : 108828759 108828891 108833624 108844316 108844478
THJ : 104800647 104803198 104818384 104818385
CPAP : 110817097 110817942 110819226
CIT : 102611429 102611728 102620382 102629675
PVY : 116107564 116109788 116126778 116126779 116126794 116126803 116129752
MINC : 123193379 123195825 123214457 123214779 123217471 123217472 123217473 123218157
TCC : 18587492 18595303 18596902 18604425 18608411 18608418
GRA : 105767480 105776955 105777054 105780579 105784022 105795004
GHI : 107888128 107892679 107894023 107897936 107898886 107907685 107908998 107915254 107916269 107918279 107937743 107942789 107944782 107944896 107944910 107949218 121203580
GAB : 108455123 108457914 108464386 108474983 108487221 108489303
HSYR : 120136887 120149580 120150788 120178429 120186546 120188411 120209578 120210532
DZI : 111274247 111281622 111281926 111282053 111293625 111308381 111309183 111310037 111315863 111315867 111318759
EGR : 104416280 104420997 104421185 104441048 104454039 104454456 104455774 108954993 120287648 120288954 120289132
GMX : 100784072 100789787 100797088 100804260 100804290 100806804 100812330 100815153 100815682 547845
GSJ : 114368711 114369300 114371778 114390958 114391170 114394888 114402919 114415698 114425929
PVU : PHAVU_004G135800g PHAVU_008G081100g PHAVU_009G142100g PHAVU_010G0584001
VRA : 106759200 106762471 106769890
VAR : 108321332 108328830 108332018 108344724 108347241
VUN : 114164233 114180361 114183116
VUM : 124825704 124828891 124835667 124835668
CCAJ : 109789540 109795015 109796020 109797526 109803572
APRC : 113850836 113862632 113867112 113869557
MTR : 11417650 25484539 25484541 25489841 25496906 25496908 25498854 25498855
TPRA : 123889054 123890013 123892852 123907936 123907945 123908537 123909610 123913430
CAM : 101502309 101503593 101512744 101513035
PSAT : 127080669 127127280 127132062
VVO : 131612039 131641132 131653283 131661186
LJA : 130710376 130718096 130718784 130721247 130728098 130735296 130738530
ADU : 107469281 107470214 107485338 107489861
AIP : 107622982 107623650 107639718 107641437
AHF : 112715285 112715805 112743867 112751575 112783457 112784376 112797237 112801794
LANG : 109325093 109337291 109343439 109352690 109358516
PCIN : 129291503 129291643 129292215 129298415 129303137 129305449 129306658 129319284
QSA : O6P43_005418 O6P43_008348 O6P43_013402 O6P43_013410 O6P43_014833 O6P43_032241
FVE : 101296053 101301008 101301724 101303508
RCN : 112169051 112185187 112197360 112199071
PPER : 18778255 18787190 18788797
PMUM : 103321124 103321125 103324675 103332884
PAVI : 110745760 110763891 110763907 110770867
PDUL : 117617424 117624656 117635349
MDM : 103419840 103432910 103434744 103435060 103437970 103444612 103447719 103451430
MSYL : 126596076 126609006 126623073 126624830 126624831 126627739
PXB : 103926602 103942444 103942807 103943123 103943279 103953750 103953796
ZJU : 107414011 107419604 107421976 107423214
MNT : 21387791 21394497 21395362 21405832
CSAV : 115706200 115708827 115723292
CSV : 101210778(GAL3) 101211924 101212167
CMO : 103488793 103488794 103496737
BHJ : 120080885 120081461 120081677
MCHA : 111010230 111010280 111015913 111015929
CMAX : 111466509 111466515 111483287 111484225
CMOS : 111447939 111447942 111449362 111449899 111450037
CPEP : 111792234 111801692 111801819 111804185
RCU : 8259536 8260752 8260754 8260757 8278538 8286475
MEAN : 126661069 126665825 126683033 126683071
JCU : 105631145 105631150 105631197 105649192 105649500
HBR : 110648108 110654552 110661560 110666614 110668068 110669696
MESC : 110603372 110614055 110619340 110624914 110628129
POP : 18107910 7455227 7455509 7459809 7490809
PEU : 105115470 105117687 105120865 105128085 105135547 105141150
PALZ : 118028868 118029057 118029058 118047349 118060222
JRE : 108980510 108991875 108995866 109012203 109012244 109019945 118348969
CILL : 122277415 122278173 122292644 122296470 122298903 122299426
CAVE : 132183737 132185645 132186055
QSU : 111998944 112022569 112023047 112028760
QLO : 115957789 115963992 115966255 115966530 115982162 115982309 115988403
TWL : 119985404 119998233 120004109 120012523
VVI : 100245638 100248541 100261154 100263458 100267960
VRI : 117922849 117923413 117924105 117924237 117928172 117928501
SLY : 101249542 101257269 778277(AGAL)
SPEN : 107012552 107015933 107021283
SOT : 102582623 102601663 102601840
SSTN : 125854607 125859634 125872101
SDUL : 129871411 129896337 129899169
CANN : 107862757 107870414 107873305
LBB : 132611307 132614071 132614072 132632456
NTA : 107769425 107770900 107779897 107793428 107798945 107801489 107830401 107831499 107831502
NSY : 104212628 104214972 104232642 104248397
NTO : 104099977 104101180 104113796 104113799
NAU : 109210862 109211580 109216005 109243492
INI : 109159506 109189689 109193709 109193959
ITR : 115996433 115997572 115998940 116009658
SIND : 105156146 105157383 105160175 105160803 105160805 105166991
OEU : 111373614 111386601 111397171 111399050 111401756 111405919 111406043
EGT : 105954276 105966143 105968238 105974988 105977584
SSPL : 121744484 121754182 121759716 121760935 121761809 121764846 121764868 121767652 121770714 121801966 121806604
SMIL : 130992996 130993235 130997173 130997176 131004973 131008330 131008343
SHIS : 125190691 125193401 125202684 125215427 125216425 125220176
APAN : 127245017 127255189 127256471 127256473 127259543 127263502
HAN : 110889244 110912778 110922858
ECAD : 122580685 122586956 122606147
LSV : 111884413 111885362 111885384 111916325
CCAV : 112508810 112509382 112527372
DCR : 108200220 108211888 108215094 108224678
CSIN : 114257936 114275091 114275092 114290167 114309604 114320631
RVL : 131299934 131299985 131301537 131320629
AEW : 130749957 130763262 130765112 130769885 130792314
BVG : 104886631 104886632 104887650
SOE : 110781962 110782120 110801557 110801714
CQI : 110702784 110704478 110716274 110716307 110723176 110730743
ATRI : 130814717 130817633 130828802
MOF : 131152831 131153707 131157929 131165518
NNU : 104588902 104591274 104593786 104594512 104610143 104610144 104610165
MING : 122064997 122069227 122079295 122079296
TSS : 122652994 122652996 122656724
PSOM : 113283717 113299660 113299661 113303523 113303525 113357833
OSA : 4344290 4348984 4348988 9269328
DOSA : Os07t0452100-00(Os07g0452100) Os07t0679300-01(Os07g0679300) Os10t0492900-01(Os10g0492900) Os10t0493600-01(Os10g0493600)
OBR : 102702220 102705647 102708369 102708454
OGL : 127752534 127779796 127779837 127786261
BDI : 100829566 100831151 100831756
ATS : 109731828 109739953 109740943 109779920 109780562
TDC : 119270187 119270198 119296811 119296812 119301191 119305014 119305015 119308730 119334252 119334274 119353282 119362405
TAES : 123043688 123046929 123046940 123051556 123101993 123103410 123106337 123111605 123115440 123115441 123120973 123122390 123122406 123124106 123181168 123181169 123187481
TUA : 125506939 125509016 125509031 125509036 125514041 125529305 125529677
LPER : 127301916 127301976 127301986 127303726 127322487 127331507 127333586
LRD : 124672501 124672989 124674140 124705701 124705711 124705714
SBI : 8057958 8063464 8063465 8063467 8065820 8065821 8074448
ZMA : 100191564 100191764 100274554 100280970 103648849
SITA : 101756166 101778063 101783050 101784704 101786040
SVS : 117835963 117838494 117843546 117843547 117845115 117845185
PVIR : 120647801 120650661 120650662 120659916 120660170 120662177 120662512 120689820 120694852 120695248 120695249
PHAI : 112874737 112875340 112880181 112880952 112881715
PDA : 103714798 103714800 103720407 120107651
EGU : 105035858 105035979 105057194
MUS : 103972718 103974157 103975852 103982384 103983387 103985882 103987929 103996513
ZOF : 121969691 121977440 121978074 121990635 121991494 121992744 121995388 121995389 121996707 122019750 122021498 122023496 122024153 122025371 122036989
DCT : 110094537 110108280 110115543
PEQ : 110017818 110021241 110021966
MSIN : 131229606 131241966 131242864 131242865 131244323 131244418 131244423 131253576 131257339 131257892
NCOL : 116248137 116262350 116264014
ATR : 18431500 18433558 18438394 18438395
SMO : SELMODRAFT_108242 SELMODRAFT_112569 SELMODRAFT_163627 SELMODRAFT_180602 SELMODRAFT_232618 SELMODRAFT_446636 SELMODRAFT_78029 SELMODRAFT_78263 SELMODRAFT_78329 SELMODRAFT_83268 SELMODRAFT_85032 SELMODRAFT_86208
CSL : COCSUDRAFT_16109 COCSUDRAFT_16143 COCSUDRAFT_17318 COCSUDRAFT_27646
CVR : CHLNCDRAFT_137182 CHLNCDRAFT_26028 CHLNCDRAFT_35902 CHLNCDRAFT_8622
TDL : TDEL_0E00170(TDEL0E00170)
PGU : PGUG_04594 PGUG_04883
PAN : PODANSg2195 PODANSg7693
PBEL : QC761_506680 QC761_607390
PPSD : QC762_506680 QC762_607390
PPSP : QC763_506680 QC763_607390
PPSA : QC764_506680 QC764_607390
MTM : MYCTH_2119446 MYCTH_2313137
MGR : MGG_09805 MGG_11374 MGG_13626 MGG_16425
PPEI : PpBr36_00001 PpBr36_00328 PpBr36_05103 PpBr36_10213
PGRI : PgNI_04769 PgNI_07475 PgNI_12038 PgNI_12488
FGR : FGSG_00068 FGSG_01803 FGSG_02059 FGSG_11169
FPU : FPSE_02844 FPSE_05150 FPSE_11350 FPSE_12030
FPOA : FPOAC1_000022(AGL2_1) FPOAC1_001902(AGL2_2) FPOAC1_002202 FPOAC1_009905
FVN : FVRRES_00060 FVRRES_02143 FVRRES_02440 FVRRES_10563
FVR : FVEG_00004 FVEG_07518 FVEG_07716 FVEG_09872 FVEG_12289 FVEG_13077
FOX : FOXG_02677 FOXG_04398 FOXG_04597 FOXG_10939 FOXG_13239 FOXG_17608
NHE : NECHADRAFT_102022 NECHADRAFT_78539 NECHADRAFT_87127
FFC : NCS54_00194600 NCS54_00469200 NCS54_01328200 NCS54_01384400
FKR : NCS57_00475400 NCS57_01318200 NCS57_01365700 NCS57_01473400
FMU : J7337_000090 J7337_001903(AGL2_1) J7337_007633 J7337_010262(AGL2_2) J7337_010477 J7337_013837
TRE : TRIREDRAFT_124016 TRIREDRAFT_27219 TRIREDRAFT_55999 TRIREDRAFT_65986 TRIREDRAFT_72632 TRIREDRAFT_75015
TRR : M419DRAFT_12566 M419DRAFT_32557 M419DRAFT_58886 M419DRAFT_6433 M419DRAFT_71638 M419DRAFT_84246 M419DRAFT_91133
TATV : 25776545(TrAtP1_011411) 25777623(TrAtP1_010689) 25777827(TrAtP1_005776) 25784997(TrAtP1_007697) 25785048(TrAtP1_012356) 25785685(TrAtP1_010724) 25785772(TrAtP1_002692)
MAW : 19249447(J3458_016491) 19249873(AGL2) 19253190(J3458_019181)
MAJ : MAA_04869 MAA_05000 MAA_09630
MBRN : 26242143(G6M90_00g046780) 26242275(agl2) 26243381(agl3) 26246960(aglA)
PCHM : VFPPC_05335 VFPPC_06917 VFPPC_07726 VFPPC_07836 VFPPC_09629 VFPPC_09630 VFPPC_10790 VFPPC_11066 VFPPC_11069 VFPPC_11915 VFPPC_13053
AMUS : LMH87_001228 LMH87_002162 LMH87_005690 LMH87_009304 LMH87_010952 LMH87_011107
PLJ : 28883894(PLICBS_005194) 28885051(PLICBS_006224) 28885053(PLICBS_006225) 28892345(PLICBS_000196) 28892856(PLICBS_009021)
PTKZ : JDV02_003417 JDV02_004097(AGL2) JDV02_005706 JDV02_008335 JDV02_008337
VAL : VDBG_02434 VDBG_05826 VDBG_06077 VDBG_07493 VDBG_07651 VDBG_09523
VDA : VDAG_02274 VDAG_02907 VDAG_03236 VDAG_04757 VDAG_09373
CFJ : CFIO01_01093 CFIO01_02759 CFIO01_03604 CFIO01_09179 CFIO01_11828 CFIO01_12353
CLUP : CLUP02_11329 CLUP02_15583
CHIG : CH63R_03489 CH63R_03847 CH63R_05312 CH63R_06271
ELA : UCREL1_11714 UCREL1_2616 UCREL1_2743 UCREL1_3832 UCREL1_5451
PFY : PFICI_00233 PFICI_00666 PFICI_04098 PFICI_09716 PFICI_12024 PFICI_13821 PFICI_14949 PFICI_15134
SSL : SS1G_01799 SS1G_03386 SS1G_04662
BFU : BCIN_01g08110 BCIN_03g02710 BCIN_08g02120
MBE : MBM_03164 MBM_03746 MBM_05577
PSCO : LY89DRAFT_617769 LY89DRAFT_643111 LY89DRAFT_659201 LY89DRAFT_682851 LY89DRAFT_695206 LY89DRAFT_696404 LY89DRAFT_753293 LY89DRAFT_764625
GLZ : GLAREA_00060 GLAREA_05542 GLAREA_07717 GLAREA_09373 GLAREA_09964
ANI : ANIA_07152 ANIA_07624 ANIA_08138 ANIA_08392 ANIA_09035
AFM : AFUA_1G01200 AFUA_4G03585 AFUA_5G02130 AFUA_5G13830 AFUA_6G02560 AFUA_8G01100 AFUA_8G01130
ACT : ACLA_003130 ACLA_016820 ACLA_044560 ACLA_044620
NFI : NFIA_023390 NFIA_029860 NFIA_039990 NFIA_048850 NFIA_073100 NFIA_094540 NFIA_094580
AOR : AO090003001305 AO090005000217 AO090010000684 AO090011000063 AO090023000151
ANG : An01g01320 An02g11150(aglB) An06g00170(aglA) An09g00260(aglC) An11g06330 An14g01800
AFV : AFLA_000374 AFLA_003000 AFLA_006516 AFLA_007422 AFLA_014177
ALUC : AKAW2_10941S AKAW2_20324S AKAW2_40025S AKAW2_40872A AKAW2_52023A
APUU : APUU_12367S APUU_22116S APUU_40024A APUU_60850A APUU_61349A APUU_70152A APUU_80289A APUU_80761S APUU_81080S
PCS : N7525_006509 N7525_008026
POU : POX_e06536 POX_f07538
TMF : EYB26_004163 EYB26_006888
TRG : TRUGW13939_04927 TRUGW13939_07520 TRUGW13939_10842 TRUGW13939_11877
PNO : SNOG_00615 SNOG_06073 SNOG_14716 SNOG_14799
PTE : PTT_04849 PTT_14879 PTT_16647 PTT_19548 PTT_20251
PTRR : 6338500(PtrM4_005560) 6342084(PtrM4_094610) 6345053(PtrM4_053440) 6347795(PtrM4_127360) 6349402(PtrM4_073140)
BZE : COCCADRAFT_36823 COCCADRAFT_4781 COCCADRAFT_8389 COCCADRAFT_97889
BSC : COCSADRAFT_134014 COCSADRAFT_136317 COCSADRAFT_174714 COCSADRAFT_27614
BOR : COCMIDRAFT_36155 COCMIDRAFT_38291 COCMIDRAFT_84676 COCMIDRAFT_98576
AALT : CC77DRAFT_1029668 CC77DRAFT_211961 CC77DRAFT_640780 CC77DRAFT_693878
ARAB : EKO05_0003321 EKO05_0004850 EKO05_0006116 EKO05_0007915
ZTR : MYCGRDRAFT_107208(MgMEL1) MYCGRDRAFT_110289(GHY)
PFJ : MYCFIDRAFT_156284 MYCFIDRAFT_185343
FFU : CLAFUR5_05373 CLAFUR5_09986
CBET : CB0940_03719 CB0940_06062 CB0940_06816 CB0940_12136
BCOM : BAUCODRAFT_145446 BAUCODRAFT_157442
NPA : UCRNP2_2238 UCRNP2_405 UCRNP2_5439 UCRNP2_7787
TASA : A1Q1_01390 A1Q1_08190
CCAC : CcaHIS019_0102320(CcaverHIS019_0102320)
PPL : POSPLDRAFT_120395 POSPLDRAFT_134790
TVS : TRAVEDRAFT_113984 TRAVEDRAFT_159574 TRAVEDRAFT_60477
DSQ : DICSQDRAFT_147615 DICSQDRAFT_148282 DICSQDRAFT_149582 DICSQDRAFT_156203 DICSQDRAFT_75292 DICSQDRAFT_79806
PCO : PHACADRAFT_210357 PHACADRAFT_247121 PHACADRAFT_251888
SHS : STEHIDRAFT_142088 STEHIDRAFT_48505 STEHIDRAFT_49883 STEHIDRAFT_53221 STEHIDRAFT_85286
HIR : HETIRDRAFT_101995 HETIRDRAFT_126780 HETIRDRAFT_39280 HETIRDRAFT_441362
PSQ : PUNSTDRAFT_120892 PUNSTDRAFT_128254 PUNSTDRAFT_131830 PUNSTDRAFT_141666 PUNSTDRAFT_64759
ADL : AURDEDRAFT_105284 AURDEDRAFT_157470
FME : FOMMEDRAFT_127614 FOMMEDRAFT_148799 FOMMEDRAFT_23758 FOMMEDRAFT_82653
GTR : GLOTRDRAFT_117566 GLOTRDRAFT_127284 GLOTRDRAFT_81761
RSX : RhiXN_02662 RhiXN_08389 RhiXN_09385 RhiXN_11608
ABP : AGABI1DRAFT106995(AGABI1DRAFT_106995) AGABI1DRAFT116817(AGABI1DRAFT_116817) AGABI1DRAFT133014(AGABI1DRAFT_133014) AGABI1DRAFT80761(AGABI1DRAFT_80761)
ABV : AGABI2DRAFT144304(AGABI2DRAFT_144304) AGABI2DRAFT194078(AGABI2DRAFT_194078) AGABI2DRAFT228269(AGABI2DRAFT_228269) AGABI2DRAFT70106(AGABI2DRAFT_70106)
MRR : Moror_13595 Moror_2709 Moror_6683 Moror_6874
MORE : E1B28_000748 E1B28_009225 E1B28_012094 E1B28_013580
SCM : SCHCO_02611511(SCHCODRAFT_02611511)
CPUT : CONPUDRAFT_138841 CONPUDRAFT_138842 CONPUDRAFT_156641
SLA : SERLADRAFT_439461 SERLADRAFT_457293 SERLADRAFT_457299
PFP : PFL1_02443 PFL1_06722
SGRA : EX895_001637 EX895_002069
PGR : PGTG_05910 PGTG_05915 PGTG_07144 PGTG_14781 PGTG_18216 PGTG_20771
PTRC : PtA15_10A650 PtA15_10A666 PtA15_11A33 PtA15_13A420 PtA15_3A75
PSTR : Pst134EA_007790 Pst134EA_009264 Pst134EA_017873 Pst134EA_017874 Pst134EA_019676 Pst134EA_019691 Pst134EA_019693
MLR : MELLADRAFT_39639 MELLADRAFT_64941 MELLADRAFT_69996 MELLADRAFT_93697
MBR : MONBRDRAFT_23493 MONBRDRAFT_28470 MONBRDRAFT_29066 MONBRDRAFT_30876 MONBRDRAFT_32350 MONBRDRAFT_34585 MONBRDRAFT_7758
SRE : PTSG_02182 PTSG_02230 PTSG_06551 PTSG_08545
DFA : DFA_08164 DFA_08423(melA) DFA_08903
ACAN : ACA1_061330 ACA1_131240 ACA1_174200 ACA1_381620
TET : TTHERM_00043770 TTHERM_01181990
SMIN : v1.2.007095.t1(symbB.v1.2.007095.t1) v1.2.007148.t1(symbB.v1.2.007148.t1) v1.2.036785.t1(symbB.v1.2.036785.t1)
AAF : AURANDRAFT_20459 AURANDRAFT_2775 AURANDRAFT_27878 AURANDRAFT_29988 AURANDRAFT_35927
EHX : EMIHUDRAFT_105406 EMIHUDRAFT_109526 EMIHUDRAFT_239510 EMIHUDRAFT_433441 EMIHUDRAFT_53809 EMIHUDRAFT_66472 EMIHUDRAFT_68712 EMIHUDRAFT_77352 EMIHUDRAFT_77441 EMIHUDRAFT_77442
GTT : GUITHDRAFT_164562 GUITHDRAFT_70739
ECOO : ECRM13514_5334(melA)
ECOH : ECRM13516_5002(melA)
ECW : EcE24377A_3380(rafA) EcE24377A_4673(melA)
EUN : UMNK88_4983 UMNK88_pK8849(rafA)
ECM : EcSMS35_3114(rafA) EcSMS35_4585(melA)
EDJ : ECDH1ME8569_3978(melA)
ECOI : ECOPMV1_04579(melA)
KPN : KPN_04203 KPN_04505(melA)
KPU : KP1_0062 KP1_0371(melA)
KPM : KPHS_00550 KPHS_03480
KPH : KPNIH24_00290 KPNIH24_01825
KPZ : KPNIH27_00310 KPNIH27_01810
KPV : KPNIH29_00260 KPNIH29_01870
KPW : KPNIH30_00295 KPNIH30_01895
KPY : KPNIH31_00295 KPNIH31_01920
KPG : KPNIH32_00295 KPNIH32_01900
KPC : KPNIH10_00290 KPNIH10_01815
KPQ : KPR0928_00290 KPR0928_01820
KPT : VK055_2961 VK055_3274(rafA)
KPO : KPN2242_00590 KPN2242_24060
KPR : KPR_0161(rafA) KPR_0486(melA)
KPI : D364_21420 D364_22950
KPS : KPNJ2_05075 KPNJ2_05350
KPX : PMK1_01693(rafA) PMK1_01979(melA)
KPB : FH42_17370 FH42_18875
KPNE : KU54_024835 KU54_026500
KPNU : LI86_24675 LI86_26275
KPNK : BN49_4653 BN49_4926(melA)
KPE : KPK_5162(melA) KPK_5478(rafA)
KPK : A593_11135 A593_12615
KVD : KR75_08540 KR75_10060
KVQ : SP68_15155 SP68_16650
KOY : J415_02695 J415_21125
KOM : HR38_05590 HR38_14960
KOK : KONIH1_00230 KONIH1_10535
KOC : AB185_06745 AB185_25665
KQU : AVR78_04865 AVR78_06420
KQV : B8P98_25670 B8P98_27320
KLL : BJF97_00315 BJF97_10785
KLW : DA718_23615 DA718_27125 DA718_29015
KAR : LGL98_23150 LGL98_24650
KGR : JJJ10_17960 JJJ10_27240
KPAS : LUW96_07050 LUW96_17120
KLC : K7H21_17740 K7H21_27465
KLM : BWI76_00605 BWI76_10320
REE : electrica_04968(rafA)
ROR : RORB6_16940 RORB6_18305
RON : TE10_02650 TE10_04165
RPLN : B1209_25005 B1209_26535 B1209_27615
RAO : DSD31_23815 DSD31_25345
RTG : NCTC13098_06765(melA) NCTC13098_07135(rafA_2)
CENS : P2W74_06140 P2W74_18930
KGO : CEW81_14790 CEW81_20445
KIE : NCTC12125_01788(melA)
LEF : LJPFL01_3086 LJPFL01_4205
BUF : D8682_21395 D8682_21405
BAGE : BADSM9389_02370 BADSM9389_07940
BFT : MNO13_05595 MNO13_05605
SUPE : P0H77_15730 P0H77_22520
EBU : CUC76_13545 CUC76_15110
YPG : YpAngola_A1681(agaN)
YPI : YpsIP31758_2395(agaN)
SRY : M621_12520 M621_15865
SFJ : SAMEA4384070_3148(melA) SAMEA4384070_4176(agaA)
SOF : NCTC11214_00827(melA) NCTC11214_03573(agaA_1)
EIC : NT01EI_1777 NT01EI_3422
ETE : ETEE_1245(melA) ETEE_3997(melA)
ETR : ETAE_1917(melA) ETAE_3011
ETC : ETAC_08755 ETAC_14505
EDL : AAZ33_09820 AAZ33_15940
EHO : A9798_02170 A9798_07525
LGU : LG3211_0296(agaA) LG3211_0298 LG3211_1843 LG3211_2557(agaA)
LYJ : FKV23_15030 FKV23_15040
RGL : CS053_01565 CS053_07755 CS053_15410
DJA : HY57_08620 HY57_08625
DCS : ISN74_00220 ISN74_09700
DTL : H8F01_16350 H8F01_19615 H8F01_19635
LRZ : BJI69_06900 BJI69_17505 BJI69_17550 BJI69_19720
LPY : FIV34_07100 FIV34_07105 FIV34_12960
LANH : KR767_00595 KR767_19130
LAEG : L2Y94_00230 L2Y94_10700 L2Y94_14875
LAEO : L2Y97_07200 L2Y97_14100
LAES : L2Y96_07755 L2Y96_13790
VVM : VVMO6_01835 VVMO6_03138
VVL : VV93_v1c12920 VV93_v1c31630
VHR : AL538_05185 AL538_15920 AL538_16220
VNA : PN96_07690 PN96_21200
VOW : A9237_02125 A9237_12750 A9237_14100 A9237_14810
VRO : BSZ04_12210 BSZ04_13650
VEJ : VEJY3_05670 VEJY3_09135 VEJY3_09535 VEJY3_21461 VEJY3_22581
VFU : vfu_A01650 vfu_B00922
VAU : VANGNB10_cI1117(galA)
VCY : IX92_02410 IX92_09895 IX92_28345
VCT : JV59_25430 JV59_28570 JV59_36295
VTU : IX91_05505 IX91_21830
VEU : IXK98_06020 IXK98_11670 IXK98_13830
VSI : MTO69_08635 MTO69_10630
VBR : A6E01_02970 A6E01_03605
VSC : VSVS12_02153(galA) VSVS12_04110(melA)
VSH : BSZ05_00485 BSZ05_12540 BSZ05_13490 BSZ05_19555
VAF : D1115_06065 D1115_09850
VAS : GT360_09155 GT360_09935 GT360_14860
VPL : SA104470976_01423(rafA)
VOS : KNV97_07265 KNV97_09705
VCRA : IS519_13995 IS519_16690
VLE : ISX51_04680 ISX51_06240 ISX51_22445
PPR : PBPRB0018(Z5721) PBPRB0258(ATU4665)
PGB : H744_1c1408 H744_2c2312
AVN : Avin_51410 Avin_51420(melA)
AVL : AvCA_51410 AvCA_51420(melA)
AVD : AvCA6_51410 AvCA6_51420(melA)
ACX : Achr_39750 Achr_39760(melA)
PART : PARC_a0217(rafA) PARC_a1715
CATE : C2869_03415 C2869_05770 C2869_07570 C2869_07730
AGZ : M0C34_03990 M0C34_06845 M0C34_11130
AGQ : LQZ07_04440 LQZ07_06020 LQZ07_14750 LQZ07_16595
AGAR : OAG1_14630 OAG1_18700 OAG1_33830(rafA)
CELL : CBR65_15165 CBR65_18810 CBR65_18855
GAI : IMCC3135_27800(rafA)
ZPL : ZBT109_1604 ZBT109_2521
MARD : IBG28_03195 IBG28_09390
GSN : YC6258_03014 YC6258_03469 YC6258_04821
RFO : REIFOR_00812 REIFOR_02177
EEI : NX720_08960 NX720_23870 NX720_25070
AEL : NCTC12917_01788(rafA)
AALL : I6G90_05690 I6G90_12335(melA)
AJD : I6H43_08540 I6H43_20730
CDIZ : CEDIAZO_00419(rafA)
BPM : BURPS1710b_A1181(rafA)
BPL : BURPS1106A_A2815(rafA)
BSTL : BBJ41_36430 BBJ41_36520
BARI : NLX30_38025 NLX30_38180
PHS : C2L64_46520 C2L64_51555
PMEG : FNZ07_05100 FNZ07_11540 FNZ07_29550
CPRA : CPter91_4517 CPter91_5272(rafA)
MALI : EYF70_07830 EYF70_07865 EYF70_13260 EYF70_16390 EYF70_18425 EYF70_19500
MUM : FCL38_02070 FCL38_08330 FCL38_08350 FCL38_10810 FCL38_10845 FCL38_29650 FCL38_30525
MFLA : GO485_02800 GO485_07220 GO485_27015
PLUE : EWM63_00635 EWM63_01955 EWM63_03440 EWM63_24945 EWM63_24985
DUG : HH213_01675 HH213_22020
TMJ : P0M04_07675 P0M04_19690 P0M04_31535
MLN : A9174_10780 A9174_12605 A9174_12630
MCI : Mesci_2357 Mesci_2362
MCIC : A4R28_28970 A4R28_28995
MOP : Mesop_2501 Mesop_2506
MAM : Mesau_02387 Mesau_02392
MAMO : A6B35_27230 A6B35_27255
MESW : A9K65_012370 A9K65_012395
MESM : EJ066_16275 EJ066_16300
MESP : C1M53_04940 C1M53_28920
MHUA : MCHK_0133(melA) MCHK_0138(melA)
MJR : EB229_12470 EB229_12495
MERD : EB233_28965 EB233_28990
PHYL : HB779_20685 HB779_20710
RBS : RHODOSMS8_00083(melA)
SME : SM_b21643(agaL2) SM_b21648(melA)
SMK : Sinme_4177 Sinme_4182
SMQ : SinmeB_4673 SinmeB_4678
SMX : SM11_pD0054(melA) SM11_pD0059(agaL2)
SMI : BN406_05140(melA1) BN406_05145(melA3)
SMEG : C770_GR4pD0055 C770_GR4pD0060
SMEL : SM2011_b21643(agaL2) SM2011_b21648(melA)
SMER : DU99_25735 DU99_25760
RHI : NGR_b02750(agaL1) NGR_b02800(agaL2)
SFH : SFHH103_06487(melA) SFHH103_06492
SFD : USDA257_c22790(melA2) USDA257_c22840(melA3)
SAME : SAMCFNEI73_pC1991(melA) SAMCFNEI73_pC1997(melA)
SINO : SS05631_b50480 SS05631_b50530
ESJ : SJ05684_b42590 SJ05684_b42640
EAK : EKH55_3976 EKH55_3981
EMX : FKV68_25295 FKV68_25320
ENU : PYH37_001806 PYH37_001811
EGI : PZN02_005503 PZN02_005508
SKM : PZL22_000766 PZL22_000771
STEG : QA637_19695 QA637_19720
EAD : OV14_b1314(agaL1) OV14_b1319(agaL2)
EAH : FA04_26780 FA04_26805
ECAA : J3R84_37105 J3R84_37130
ENP : JVX98_05520 JVX98_05545
ATU : Atu4660(melA) Atu4665(melA)
ATA : AWN88_02375 AWN88_02400
AGR : AGROH133_09618 AGROH133_09638
ATF : Ach5_43840(melA) Ach5_43890(melA)
AGT : EYD00_22520 EYD00_22545
ARUI : G6M88_18745 G6M88_18770 G6M88_21700
AVV : RvVAT039_35370 RvVAT039_35420 RvVAT039_40700
AVF : RvVAR031_33250 RvVAR031_38230 RvVAR031_38280
ASAL : CFBP5507_15720 CFBP5507_15745
RIR : BN877_II1755(melA1) BN877_II1760(melA2)
RPUS : CFBP5875_22420 CFBP5875_22445
ACUC : KZ699_23280 KZ699_23305
ALEG : CFBP4996_24645 CFBP4996_24670 CFBP4996_25815
AVI : Avi_5115(melA) Avi_5121(melA) Avi_5740
RET : RHE_PE00089(agaL1) RHE_PE00094(agaL2)
REC : RHECIAT_PA0000096(agaL1) RHECIAT_PA0000101(agaL2)
REL : REMIM1_PD00090(melA-1) REMIM1_PD00095(melA-2)
REP : IE4803_PA00099(melA-1) IE4803_PA00104(melA-2)
REI : IE4771_PC00090(melA-1) IE4771_PC00095(melA-2)
RLE : pRL110242 pRL110247 pRL120256(melA)
RLT : Rleg2_4755 Rleg2_5903 Rleg2_5908
RLG : Rleg_5368 Rleg_6873 Rleg_6878
RLB : RLEG3_06465 RLEG3_06490 RLEG3_09900
RLU : RLEG12_03260 RLEG12_03285
RTR : RTCIAT899_PC07200 RTCIAT899_PC07225
RHL : LPU83_pLPU83d1843(melA1) LPU83_pLPU83d1848(melA3)
RGA : RGR602_PC00727(melA-1) RGR602_PC00732(melA-2)
RHN : AMJ98_PD00104(agaL-1) AMJ98_PD00109(agaL-2)
RPHA : AMC79_PB00093(agaL-1) AMC79_PB00098(agaL-2)
RHX : AMK02_PE00105(agaL-1) AMK02_PE00110(agaL-2)
RHV : BA939_17515 BA939_17540
RHK : Kim5_PB00103(agaL-1) Kim5_PB00108(agaL-2)
REZ : AMJ99_PC00104(agaL-1) AMJ99_PC00109(agaL-2)
RJG : CCGE525_29675 CCGE525_29700
RHR : CKA34_23780 CKA34_23805 CKA34_32865
RGR : FZ934_21065 FZ934_21090
RAD : CO657_31435 CO657_31460
ROY : G3A56_25140 G3A56_25165
RII : FFM53_025450 FFM53_031525 FFM53_031550
RHID : FFM81_031025 FFM81_031050
RBQ : J2J99_27495 J2J99_27520
RLN : J0663_28085 J0663_28110
RBAN : J2J98_24435 J2J98_24460
RRG : J3P73_26205 J3P73_29340 J3P73_29365
RLS : HB780_05190 HB780_05215 HB780_10825
RAW : NE851_03200 NE851_03225
RSUL : N2599_22715 N2599_22740
RLW : RlegWSM1455_24800 RlegWSM1455_29945 RlegWSM1455_29970
RRHO : PR018_27580 PR018_27605
RTU : PR017_26310 PR017_26335
ARA : Arad_8317 Arad_9538(agaL2) Arad_9545(agaL1)
ARO : B0909_14180 B0909_14205
RBW : RLCC275e_24525 RLCC275e_28910 RLCC275e_28935
RHIZ : QMO80_005335 QMO80_005340
NGL : RG1141_PA14650 RG1141_PA14700
NGG : RG540_PA16350 RG540_PA16400
NPM : QEO92_05800 QEO92_05825
KAI : K32_38900(galA) K32_40480(melA) K32_40530
LNE : FZC33_06105 FZC33_23095
MOC : BB934_41845 BB934_43310 BB934_43350
DEQ : XM25_00070 XM25_00350
DEI : C4375_01820 C4375_13415
DEA : FPZ08_10165 FPZ08_10175
DSAL : K1X15_04580 K1X15_14385
DEVO : H4N61_03515 H4N61_12865
DNP : N8A98_14615 N8A98_14620
DOY : JI749_07605 JI749_07615
DAG : PSQ19_02350 PSQ19_02385 PSQ19_09380
DRP : PSQ90_11800 PSQ90_11810 PSQ90_14875
YTI : FNA67_04095 FNA67_04440
MEY : TM49_11570 TM49_22025
MAAD : AZF01_02505 AZF01_02530
MMED : Mame_00438(melA_1) Mame_00443(melA_2) Mame_03624(melA_3)
MLUT : JET14_03555 JET14_03580
AUA : M673_01610 M673_01620
RPOP : K1718_05825 K1718_06180
CAK : Caul_0313 Caul_0315 Caul_2129 Caul_2412
CFH : C1707_05555 C1707_15790
BFQ : JX001_14355 JX001_14365
AEX : Astex_1018 Astex_2589 Astex_3319 Astex_3509
ASTI : Q1W73_00430 Q1W73_07800 Q1W73_13740
ASTD : ATDW_04660 ATDW_08150 ATDW_22660
TSV : DSM104635_01779(rafA)
RLI : RLO149_c015760(rafA)
OTM : OSB_25540(melA) OSB_28060(rafA)
AHT : ANTHELSMS3_01218(rafA)
LVS : LOKVESSMR4R_02334(rafA)
LIT : FPZ52_14245 FPZ52_14250
PPAF : I8N54_07810 I8N54_18830 I8N54_18840
NSM : JO391_13490 JO391_13505
AALK : LGT41_0005335 LGT41_0011045
BOSG : GKR98_02360(melA) GKR98_10410
NOV : TQ38_011825 TQ38_024030 TQ38_026465
NOT : C7W88_12265 C7W88_19825
NCP : U0041_12290 U0041_20345
STAX : MC45_01240 MC45_01250 MC45_05755 MC45_08925 MC45_12665
SSAN : NX02_07590 NX02_16900
SPAN : AWL63_00275 AWL63_05190 AWL63_16455
SPLM : BXU08_00225 BXU08_08675 BXU08_16575 BXU08_16595
SPLK : AV944_06295 AV944_13440
SPAU : DRN02_002185 DRN02_007290
SPAP : H3Z74_00375 H3Z74_13265 H3Z74_23435
SBIN : SBA_ch1_18150 SBA_ch2_7590
SMOR : LHA26_11385 LHA26_11395
SCAB : LZK98_08525 LZK98_16680
SNOS : K8P63_12095 K8P63_13700
SSAG : KV697_00230 KV697_03155 KV697_04660
SPHR : BSY17_1069 BSY17_3561
SYA : A6768_10900 A6768_12780 A6768_14170
SPYG : YGS_C1P1482 YGS_C2P0907 YGS_C2P1185
SAMI : SAMIE_1002470 SAMIE_1002550 SAMIE_1034070
SPHS : ETR14_01445 ETR14_08060 ETR14_12060 ETR14_17925 ETR14_17945
SAND : H3309_07120 H3309_08420
ADO : A6F68_00806(agaA_1) A6F68_00827(agaA_2) A6F68_01126(rafA)
CMAN : A9D14_04555 A9D14_16550
PHZ : CHX26_04755 CHX26_05085
LCK : HN018_11340 HN018_17715
NAO : Y958_02975 Y958_16115 Y958_20885 Y958_23760
BAZ : BAMTA208_14810(melA)
BACQ : DOE78_04305 DOE78_11885 DOE78_22495
BACA : FAY30_03260 FAY30_26880
BARD : QRY57_13395 QRY57_13400
BCO : Bcell_1099 Bcell_1103 Bcell_1453
BCIR : C2I06_14825 C2I06_16200
NTX : NQZ71_05485 NQZ71_17065 NQZ71_20145 NQZ71_25110 NQZ71_25120
NIL : L8T27_004895 L8T27_016180 L8T27_020225 L8T27_025550
BMQ : BMQ_0457 BMQ_2753(melA)
BMD : BMD_0459 BMD_2737(melA)
BMH : BMWSH_2447(melA) BMWSH_4786(galA)
BMEG : BG04_2750 BG04_5240
BHA : BH1870 BH2223 BH2228(melA)
OON : NP440_03310 NP440_03340
GYC : GYMC61_0528 GYMC61_2733
GYA : GYMC52_1863 GYMC52_2132
GEJ : A0V43_06915 A0V43_08755
GZA : IC807_05745 IC807_06915
PCAL : BV455_01546(rafA_1) BV455_03267(rafA_2)
AXL : AXY_05510(aga) AXY_13780 AXY_14140(melA) AXY_21480
HMN : HM131_04500 HM131_04520
TAP : GZ22_02375 GZ22_13680
PPSR : I6J18_05240 I6J18_08825
RAX : KO561_15925 KO561_16585
PRD : F7984_12365 F7984_12625 F7984_16645
GRC : GI584_07490 GI584_21420 GI584_21445(melA) GI584_22055
GCS : MUN88_08805 MUN88_08810 MUN88_20910
GSM : MUN87_10060 MUN87_10065 MUN87_20700
NDT : L1999_02815 L1999_04035 L1999_09425 L1999_10825 L1999_14290 L1999_24505 L1999_26925
NCM : QNK12_01965 QNK12_02095
NNV : QNH39_03395 QNH39_25755(melA)
MEKU : HUW50_00215 HUW50_07020 HUW50_14730 HUW50_15310 HUW50_17300 HUW50_17640
MSEM : GMB29_03530 GMB29_15475(melA)
MDG : K8L98_01900 K8L98_15215 K8L98_20530
MLIT : KDJ21_024765 KDJ21_025520
MEBZ : LPC09_05610 LPC09_22915
BLEN : NCTC4824_03506(melA_1)
SLMS : MM221_05545 MM221_05885 MM221_05930 MM221_05960 MM221_14335
ALKL : MM271_05760 MM271_06675 MM271_17860
DOM : RRU94_05390 RRU94_24625
AHAL : FTX54_001545 FTX54_001570
EXU : ESP131_04375 ESP131_09500
EACE : KKI46_02190 KKI46_07290
BBE : BBR47_28110(melA) BBR47_28160
PJD : Pjdr2_0045 Pjdr2_0390 Pjdr2_1293 Pjdr2_1299 Pjdr2_3202 Pjdr2_3562 Pjdr2_4264 Pjdr2_5058 Pjdr2_5454
GYM : GYMC10_1358 GYMC10_4397 GYMC10_4405 GYMC10_4406 GYMC10_5601
PPY : PPE_00551 PPE_00697 PPE_02216 PPE_02329
PPM : PPSC2_03045(melA1) PPSC2_03910(melA3) PPSC2_05095(melA5) PPSC2_11525 PPSC2_12010(melA9) PPSC2_12910(rafA)
PPO : PPM_0551(melA1) PPM_0727(melA3) PPM_0958(melA5) PPM_2208(M1_2499) PPM_2298(melA9) PPM_2475(rafA)
PPOL : X809_02520 X809_03445 X809_11935 X809_27945
PPQ : PPSQR21_005860(galA) PPSQR21_007560 PPSQR21_010120(celF) PPSQR21_022940 PPSQR21_023880 PPSQR21_025620
PPOY : RE92_00345 RE92_00805 RE92_06835 RE92_08075 RE92_08890 RE92_23875
PMS : KNP414_01552(melA2) KNP414_04679 KNP414_06099 KNP414_06343
PMQ : PM3016_1859 PM3016_4088 PM3016_5729 PM3016_5955
PMW : B2K_09445 B2K_18925 B2K_21230 B2K_29170 B2K_30275
PTA : HPL003_12895 HPL003_22225 HPL003_22715
PSAB : PSAB_03840 PSAB_12220
PDU : PDUR_04380 PDUR_13880
PBD : PBOR_04740 PBOR_08220 PBOR_09605 PBOR_11385 PBOR_13685 PBOR_17410 PBOR_18455 PBOR_25820 PBOR_28815
PGM : PGRAT_08050 PGRAT_08285 PGRAT_08350 PGRAT_10535 PGRAT_15210 PGRAT_16605 PGRAT_23050 PGRAT_23715 PGRAT_27685
POD : PODO_04255 PODO_07665 PODO_09090 PODO_16050 PODO_22510 PODO_22580
PAEN : P40081_05020 P40081_08255 P40081_08420 P40081_11150 P40081_13505 P40081_17765 P40081_18800 P40081_25970
PAEF : R50345_04095 R50345_07770 R50345_16270 R50345_22380
PAEQ : R50912_07385 R50912_08750 R50912_10635 R50912_13110 R50912_16560 R50912_17430 R50912_23945
PSTE : PSTEL_04700 PSTEL_08135 PSTEL_09880 PSTEL_15515
PAEA : R70723_03875 R70723_08015 R70723_08225 R70723_10405 R70723_12535 R70723_24125 R70723_24150 R70723_29030
PAEE : R70331_03890 R70331_07515 R70331_07540 R70331_08060 R70331_08265 R70331_10050 R70331_11980 R70331_28020
PAEH : H70357_04460 H70357_08445 H70357_10880 H70357_14330 H70357_14455 H70357_19280 H70357_20925 H70357_29805 H70357_33375
PAEJ : H70737_07485 H70737_08170 H70737_15775 H70737_22690 H70737_22745
PBJ : VN24_00665 VN24_06705 VN24_13130 VN24_22595
PRI : PRIO_1704(agaR1) PRIO_1726 PRIO_1774(agaR3) PRIO_2291 PRIO_2637 PRIO_3323 PRIO_3664(melA3) PRIO_5005 PRIO_5329 PRIO_5785(agaR5)
PSON : JI735_03800 JI735_16795 JI735_17145 JI735_20260 JI735_24080 JI735_24620 JI735_25405 JI735_32625 JI735_33180
PPEO : ABE82_02755 ABE82_03640 ABE82_05000 ABE82_11720 ABE82_12245
POW : IJ21_06940 IJ21_10740 IJ21_10790 IJ21_36240 IJ21_44870
PBV : AR543_05840 AR543_15810
PXL : BS614_04710 BS614_07785 BS614_10790 BS614_18535
PYG : AWM70_01055 AWM70_05895 AWM70_06840 AWM70_06915 AWM70_06920 AWM70_07895 AWM70_10510 AWM70_14045
PSWU : SY83_02135 SY83_02175 SY83_03520 SY83_04355 SY83_04405 SY83_04410 SY83_08070 SY83_09285 SY83_12130 SY83_20330 SY83_20880 SY83_22450
PDH : B9T62_01920 B9T62_02115 B9T62_05030 B9T62_08730 B9T62_13540 B9T62_17560 B9T62_25245 B9T62_37460
PIB : BBD41_07995 BBD41_18080 BBD41_21145 BBD41_28300
PCX : LPB68_08250 LPB68_17555 LPB68_18725 LPB68_18805 LPB68_21145
PKB : B4V02_12110 B4V02_13565 B4V02_16090 B4V02_20240
PAIH : ASL14_06720 ASL14_23965 ASL14_24610
PVO : PVOR_08855 PVOR_23949 PVOR_24024 PVOR_24029
PLW : D5F53_01970 D5F53_08155 D5F53_08160 D5F53_08205 D5F53_22815
PLEN : EIM92_02805 EIM92_05405 EIM92_18270
PPSC : EHS13_02385 EHS13_03170 EHS13_04875 EHS13_04880 EHS13_06495 EHS13_07195 EHS13_09195 EHS13_12065 EHS13_14790 EHS13_17005 EHS13_21335 EHS13_21410 EHS13_22105 EHS13_23265 EHS13_24220 EHS13_25665 EHS13_28325 EHS13_30925 EHS13_33380 EHS13_35850
PLUT : EI981_01475 EI981_16675 EI981_18220 EI981_22875
PALB : EJC50_00465 EJC50_10230 EJC50_16450 EJC50_18665 EJC50_19385 EJC50_23645 EJC50_23705 EJC50_26835 EJC50_28165
PCHI : PC41400_09705 PC41400_18090
PBK : Back11_02100 Back11_04490(melA) Back11_15520 Back11_19070 Back11_33780 Back11_55350
PBAC : HUB98_01360 HUB98_18400 HUB98_21280 HUB98_23545
PRZ : GZH47_11505(melA) GZH47_13915 GZH47_20170(melA) GZH47_25240 GZH47_27100 GZH47_27155 GZH47_32010
PLYC : GXP70_01350 GXP70_02765 GXP70_05245(melA) GXP70_20490 GXP70_20545 GXP70_23005 GXP70_23980 GXP70_24420(melA) GXP70_26455(melA)
PSOP : KP014_07220 KP014_11350 KP014_17030
PTRI : KDC22_07185 KDC22_07405 KDC22_09745 KDC22_12190 KDC22_16425 KDC22_22605
PALO : E6C60_0461 E6C60_3598
PCEL : HUB94_09175 HUB94_12405 HUB94_25565 HUB94_25880 HUB94_26745
PTJ : JRJ22_03410 JRJ22_06590 JRJ22_08595 JRJ22_10475 JRJ22_24885 JRJ22_27575
PALR : HGI30_05135 HGI30_08340
PKA : PQ456_01095 PQ456_03280 PQ456_12040(melA) PQ456_19450
PUI : PUW25_05905 PUW25_19455 PUW25_24345
PMAH : PTQ21_06495 PTQ21_11940 PTQ21_15675 PTQ21_15695 PTQ21_20660(melA)
PAMY : P9222_00490 P9222_04005 P9222_22715
PDY : QJQ58_00145 QJQ58_02925
PMAE : LMZ02_03005 LMZ02_03930(melA) LMZ02_16730 LMZ02_24040 LMZ02_28320
PWN : QNH46_02150 QNH46_10430 QNH46_23050
PSPN : L1F29_01645 L1F29_16345 L1F29_18200 L1F29_22570 L1F29_24360(melA)
PPOG : QPK24_03325 QPK24_04510 QPK24_05350 QPK24_07970 QPK24_09790 QPK24_20825 QPK24_23175
PAUN : MJA45_08705 MJA45_19580 MJA45_23820
PHK : SK066_03895 SK066_12770
PNK : AASFL403_06925 AASFL403_15895 AASFL403_17835 AASFL403_22230
PPAB : KET34_01265 KET34_04295 KET34_07400 KET34_11795 KET34_11815 KET34_16530(melA)
PKP : SK3146_00915(melA_1) SK3146_01760(rafA_1) SK3146_01764(rafA_2) SK3146_02043(melA_2)
PGLU : A3958_00410 A3958_00460 A3958_00465 A3958_15360
PHEX : L0M14_15655 L0M14_16245
TCO : Theco_0425 Theco_0500 Theco_1500
COH : EAV92_00020 EAV92_00050 EAV92_00055 EAV92_07085 EAV92_10035 EAV92_13265 EAV92_15675
COHN : KCTCHS21_05810 KCTCHS21_08150 KCTCHS21_09060 KCTCHS21_09410 KCTCHS21_09810 KCTCHS21_10450(melA) KCTCHS21_20820(galA) KCTCHS21_21380 KCTCHS21_21640 KCTCHS21_24260 KCTCHS21_48890
CCHL : FPL14_12535 FPL14_19235 FPL14_22565 FPL14_25780
CHEB : HH215_02915 HH215_05760 HH215_15630 HH215_21395 HH215_29980
COHL : J4772_09980 J4772_19215(melA) J4772_21015(melA) J4772_21090 J4772_22010 J4772_23130 J4772_24105 J4772_28480
SACA : FFV09_05360 FFV09_07355 FFV09_09050 FFV09_12155 FFV09_12405
PLIG : NAG76_11550 NAG76_14855 NAG76_17075
ACUR : JZ785_10160 JZ785_22585 JZ785_25170
ADAU : NZD86_18860 NZD86_23160
FFE : LSG31_11420 LSG31_12060
VIJ : JNUCC6_21770 JNUCC6_21795
NTR : B0W44_05980 B0W44_09630
LCAR : BHS00_03745 BHS00_04645
SPW : SPCG_1872(aga) SPCG_2129
SNV : SPNINV200_17190(aga)
SNM : SP70585_1956 SP70585_2287
SNB : SP670_1974 SP670_2306
SNX : SPNOXC16690(aga) SPNOXC19060
SPNE : SPN034156_07420(aga)
SPNU : SPN034183_16720(aga) SPN034183_19090
SPNM : SPN994038_16610(aga) SPN994038_18980
SPNO : SPN994039_16620(aga) SPN994039_18990
SMC : SmuNN2025_1137(agaL)
SSB : SSUBM407_0162 SSUBM407_1450(aga)
SSU : SSU05_0172 SSU05_1560
SSV : SSU98_0174 SSU98_1567
SSI : SSU0167 SSU1373(aga)
SSS : SSUSC84_0160 SSUSC84_1403(aga)
SSF : SSUA7_0164 SSUA7_1390
SSW : SSGZ1_0159 SSGZ1_1390
SSUS : NJAUSS_0183 NJAUSS_1440
SST : SSUST3_0179 SSUST3_1400
SSUY : YB51_0855 YB51_6895
SSK : SSUD12_0167 SSUD12_1513
SSQ : SSUD9_0177 SSUD9_1551
SUI : SSUJS14_0169 SSUJS14_1525
SUO : SSU12_0168 SSU12_1503
SSUT : TL13_0215(aga) TL13_1365(aga)
SSUI : T15_0159(galA) T15_1558
SGA : GALLO_0167 GALLO_0191
SGG : SGGBAA2069_c01850(rafA) SGGBAA2069_c02090(aga) SGGBAA2069_c05330(galA)
SGT : SGGB_0211(galA1) SGGB_0235(galA2)
STB : SGPB_0154(galA.1) SGPB_0178(galA.2) SGPB_1494(galA.3)
SCP : HMPREF0833_10744 HMPREF0833_10749
SRQ : SR187_1070 SR187_3870
SMEN : SAMEA4412692_1784(rafA) SAMEA4412692_1985(galA1)
SCAI : NCTC12191_00337(rafA)
SPSU : NCTC13786_01714(rafA)
SPOC : NCTC10925_01441(rafA)
SOS : INT76_03055 INT76_05810 INT76_09320
LCR : LCRIS_01403(galA1) LCRIS_01509(galA2)
LAMY : B1745_08070 B1745_08180
LPW : LpgJCM5343_02550(galA)
LUU : H4B44_01675 H4B44_03635 H4B44_03640 H4B44_03750 H4B44_10835
LMUL : PUW59_01890 PUW59_05690
LCZ : LCAZH_0992 LCAZH_2042
LCW : BN194_11750(agaR) BN194_22160(agaS_2)
LCL : LOCK919_1193 LOCK919_2258
LPQ : AF91_03340 AF91_08715
LPI : LBPG_00778 LBPG_02001
LPAP : LBPC_0975 LBPC_2021
LCB : LCABL_12010(galA) LCABL_22590(agaS)
LCX : LCA12A_0191 LCA12A_2329
LRH : LGG_01001(galA) LGG_02079(melA)
LRL : LC705_01059(galA) LC705_02074(melA) LC705_02697(galA)
LRA : LRHK_1040 LRHK_2077 LRHK_2806
LRO : LOCK900_0969 LOCK900_2027 LOCK900_2696
LRC : LOCK908_1076 LOCK908_2141 LOCK908_2777
LCHI : KG086_04335 KG086_09110 KG086_12410
LZE : KG087_04875 KG087_09695
LHN : LHUE1_000184 LHUE1_002083
LPJ : JDM1_0174(melA) JDM1_2777(melA)
LPT : zj316_0097 zj316_0114(melA) zj316_0391(melA)
LPR : LBP_cg0159 LBP_cg2832 LBP_p2g003
LPB : SH83_00790 SH83_14435
LPX : ASU28_13865 ASU28_13950
LOR : AYI71_00880 AYI71_09735
LPAS : KZE55_01715 KZE55_09600
LBN : LBUCD034_0290 LBUCD034_2041
LPAR : FAM21731_00732 FAM21731_02646
LCU : PL11_001160 PL11_005520
LKF : DNL43_05950 DNL43_06265
LKO : ABN16_02945 ABN16_09560 ABN16_09565 ABN16_09575
LSUA : H3M12_01045 H3M12_05220
LNA : RIN67_01010 RIN67_01030 RIN67_09280
LSI : HN6_00319 HN6_00834(galA)
LSJ : LSJ_0369 LSJ_1010c(galA)
LCY : LC20004_10345 LC20004_13065
LPD : AYR62_00445 AYR62_01935
LHB : D1010_02855 D1010_08870 D1010_15130 D1010_15545 D1010_15625 D1010_16530
WJO : FOL01_0842 FOL01_0872
WHE : EFA59_01605 EFA59_04135
EFU : HMPREF0351_11597(galA2)
ECAS : ECBG_00361 ECBG_00456 ECBG_01578 ECBG_01649
EGA : AL523_04330 AL523_07400 AL523_15930 AL523_16255 AL523_16740
ESS : ATZ33_09370 ATZ33_15330
EGV : EGCR1_14795 EGCR1_15350
ECEC : NCTC12421_00664(rafA)
ERAF : J9537_15415 J9537_19600
EIE : ENLAB_06210(galA_1) ENLAB_07220(galA_2) ENLAB_17150 ENLAB_22990
ADC : FOC79_01690 FOC79_02425
ABAE : CL176_09325 CL176_09330
FCT : NRE15_03705 NRE15_05500
CAW : Q783_09840 Q783_10165
CVD : LHA31_04565 LHA31_06480 LHA31_12545
JEH : EJN90_02040 EJN90_02215 EJN90_04850 EJN90_09290 EJN90_09685
JPO : G7058_04285 G7058_09460
CBE : Cbei_4359 Cbei_4441 Cbei_4442
CBZ : Cbs_4359 Cbs_4441 Cbs_4442
CBEI : LF65_04883 LF65_04977 LF65_04978 LF65_05415 LF65_05429 LF65_05438
CCB : Clocel_0560 Clocel_2511 Clocel_2800 Clocel_3640
CLB : Clo1100_0192 Clo1100_0917 Clo1100_1931 Clo1100_4039
CSR : Cspa_c08150(agaR1) Cspa_c23080(agaR2) Cspa_c41060 Cspa_c52700(agaR3)
CBUT : ATN24_14750 ATN24_15375
CEU : A7L45_08695 A7L45_10415
CDY : F3K33_22290 F3K33_22750 F3K33_22755
CMAH : C1I91_20595 C1I91_23985
CNN : CNEO_0955 CNEO_2431 CNEO_4136
CGEL : psyc5s11_47580 psyc5s11_47600(melA) psyc5s11_47650 psyc5s11_47660
CTHD : CDO33_05125 CDO33_10445 CDO33_10450 CDO33_20275
CLOD : C820_000078(agaSK_1) C820_002156(agaSK_2) C820_002793(agaSK_3)
OCW : OW730_06030 OW730_10170
FSA : C5Q98_00260 C5Q98_04740 C5Q98_04850
RUK : A4V00_06820 A4V00_08185 A4V00_16040
CCE : Ccel_1237 Ccel_1597 Ccel_3426
RHER : EHE19_002800 EHE19_007205 EHE19_007720 EHE19_017705
RPAY : P0092_05170 P0092_11265 P0092_21665
CSS : Cst_c04430(agaR1) Cst_c16450(agaR3) Cst_c16870
CSD : Clst_0423 Clst_1586(agaA) Clst_1627
ESR : ES1_05550 ES1_05860 ES1_16690
ESU : EUS_15060 EUS_25050 EUS_25330
FPLA : A4U99_12085 A4U99_12090
EHA : Ethha_0898 Ethha_1782
RAL : Rumal_0146 Rumal_0597 Rumal_1008 Rumal_1055 Rumal_1567
RUM : CK1_15360 CK1_19020 CK1_21840 CK1_21870 CK1_38730
RUS : RBI_I01950 RBI_II00376
FPRA : CG447_03010 CG447_05440
FDU : GXM22_03505 GXM22_05880
CAML : H6X83_01565 H6X83_07160 H6X83_11040
CARL : PXC00_01945 PXC00_11400
SVJ : NQ490_02120 NQ490_05285
PALM : RBG61_05025 RBG61_09190
AMUR : ADH66_00745 ADH66_12280
ALAC : JYE50_02735 JYE50_06915 JYE50_08925
AHC : JYE49_03445 JYE49_08610 JYE49_08805
CBAF : JS518_00500 JS518_00690 JS518_06065
BPB : bpr_I0090(gh27A) bpr_I0205(aga27A) bpr_I0788(aga36A) bpr_I2883(aga36B) bpr_III065(aga36C)
BHU : bhn_I0082 bhn_I0644 bhn_I1287 bhn_I1583 bhn_I1945
RHO : RHOM_06015 RHOM_06425 RHOM_11480
RIX : RO1_10650 RO1_18030 RO1_22250 RO1_31280
RIM : ROI_10050 ROI_26950 ROI_33920 ROI_36280
RRK : H8S51_001115 H8S51_003095 H8S51_008000 H8S51_013970
COO : CCU_08830 CCU_19870 CCU_22950 CCU_23620
CCOM : I6K69_09785 I6K69_10490 I6K69_13855
CEV : LK421_03035 LK421_04805 LK421_05105 LK421_06615 LK421_09020
BYL : A4V09_07585 A4V09_19295 A4V09_19485 A4V09_19585 A4V09_21135 A4V09_22880
BLAU : DQQ01_01735 DQQ01_06945 DQQ01_14875 DQQ01_15665 DQQ01_15745
BPRO : PMF13cell1_00611(melA_1) PMF13cell1_00855(agaA) PMF13cell1_01199(melA_3) PMF13cell1_01300(rafA_1) PMF13cell1_01634(rafA_2) PMF13cell1_01656(rafA_3) PMF13cell1_02068(rafA_4) PMF13cell1_02910(rafA_5) PMF13cell1_03321(rafA_6)
BLAB : EYS05_00305 EYS05_02755 EYS05_04305 EYS05_09675 EYS05_09685
BWX : NQ550_16720 NQ550_16750
BMAS : LK422_03245 LK422_05485 LK422_14010 LK422_14020
BLAR : LC508_19145 LC508_19265 LC508_19355 LC508_21150 LC508_21515
BLUI : Blut17040_02130 Blut17040_02530 Blut17040_27290
BCOC : BLCOC_00730(agaSK) BLCOC_00940(agaA_1) BLCOC_01200(melA_2) BLCOC_19840(agaA_2) BLCOC_51530(melA_3) BLCOC_52290
CPY : Cphy_0769 Cphy_1936 Cphy_3056
LACY : A4V08_23945 A4V08_30265 A4V08_30930 A4V08_35060
CHYL : CE91St63_23790(melA)
CSH : Closa_0319 Closa_4210
LXA : OW255_05955 OW255_19285 OW255_19310
LGG : QJR73_01285 QJR73_03390
PXV : FXF36_01010 FXF36_06585 FXF36_07065 FXF36_13505
ANR : Ana3638_01260 Ana3638_05765 Ana3638_13910 Ana3638_17035(melA) Ana3638_22145 Ana3638_22800 Ana3638_24540
ACEL : acsn021_03160 acsn021_34280 acsn021_36290
ACHT : bsdcttw_11860 bsdcttw_16190 bsdcttw_24170(melA) bsdcttw_31060 bsdcttw_42020 bsdcttw_42030 bsdcttw_43080
RTO : RTO_02770 RTO_13840 RTO_25230
RGN : RGna_17195 RGna_17200
CBOL : CGC65_00640 CGC65_19745
SINT : K5I26_03250 K5I26_05970 K5I26_05990 K5I26_06920
SSUN : H9Q77_01880 H9Q77_08230
MFOR : NQ534_10920 NQ534_12245 NQ534_14495
AHB : bsdtb5_13910 bsdtb5_14590 bsdtb5_34100 bsdtb5_39680
ERE : EUBREC_0489 EUBREC_3387
KIB : RBB56_04115 RBB56_11660 RBB56_11725
CBIL : EUBC25_07010 EUBC25_07060(galA)
HHJ : NQ487_01515 NQ487_02175 NQ487_21975
CLE : Clole_3326 Clole_3366 Clole_3367
CEW : EKH84_19655 EKH84_20060 EKH84_20065
CBAR : PATL70BA_1789(agaA)
VGU : HYG85_14595 HYG85_16210 HYG85_23435
VPY : HZI73_08370 HZI73_20735 HZI73_23485 HZI73_24500
MAS : Mahau_1075 Mahau_1944 Mahau_2684 Mahau_2711 Mahau_2969
HHL : Halha_1333 Halha_2513 Halha_2519
COB : COB47_0428 COB47_1421
CHD : Calhy_1622 Calhy_2183
COW : Calow_0333 Calow_0927 Calow_1763
CKI : Calkr_1093 Calkr_1843
CLC : Calla_0522 Calla_1244
CKN : Calkro_0579 Calkro_1590 Calkro_2165
CCHA : ELD05_02445 ELD05_08625
CDIA : CaldiYA01_13940 CaldiYA01_19810
CDAN : SOJ16_000418 SOJ16_001059
MHG : MHY_17160 MHY_27260 MHY_28160
MFUN : GXM21_03335 GXM21_07200
FRO : AALO17_18490 AALO17_25860
EBM : SG0102_02850(melA) SG0102_15380
TUR : AT726_08105 AT726_08965
TSG : HLK68_07420 HLK68_08680 HLK68_08685
TBB : J0J69_11035 J0J69_12200
TFC : T23_03060(galA_1) T23_04680(galA_2)
FIT : Fi14EGH31_29210(galA)
SCHI : SCHIN_v1c09860(galA)
MDX : BTO20_06525 BTO20_08985
MLL : B1R94_22295 B1R94_27255
MAUU : NCTC10437_01374(melA)
MMAG : MMAD_16850 MMAD_41740
MAIC : MAIC_02180 MAIC_26500
MHEV : MHEL_07780 MHEL_24530 MHEL_49670
SCO : SCO0274(SCF85.02) SCO0284(SCF85.12) SCO0541(SCF11.21)
SMA : SAVERM_1082(agaB1) SAVERM_1475(agaB2) SAVERM_1641(agaB3)
SGRF : SGFS_026570 SGFS_029020 SGFS_078420
SCB : SCAB_12251 SCAB_12861(aga) SCAB_3891 SCAB_57691
SSX : SACTE_6011 SACTE_6015
SVL : Strvi_2244 Strvi_2248 Strvi_2875 Strvi_3296 Strvi_3740 Strvi_4330 Strvi_5822
SHY : SHJG_0110 SHJG_0246 SHJG_1048 SHJG_2526 SHJG_7663 SHJG_8422 SHJG_8426
SHO : SHJGH_0881 SHJGH_2290 SHJGH_7424 SHJGH_8183 SHJGH_8187
SRC : M271_02455 M271_03605 M271_06760 M271_08745 M271_11125 M271_14475 M271_14495 M271_43050
SMAL : SMALA_0788 SMALA_5861 SMALA_5865 SMALA_6773 SMALA_6832 SMALA_7647
SSOI : I1A49_00145 I1A49_07520 I1A49_33020 I1A49_33040 I1A49_37630 I1A49_37935 I1A49_42010
SBH : SBI_01330 SBI_02019 SBI_02098 SBI_02579(agaB3) SBI_07341 SBI_07398 SBI_09778 SBI_09936
SALS : SLNWT_4309 SLNWT_4446
SLV : SLIV_35235 SLIV_36495 SLIV_36545
SGU : SGLAU_26945 SGLAU_28330
SVT : SVTN_00920 SVTN_00990 SVTN_36345
SCW : TU94_29130 TU94_29600
SLC : SL103_12745 SL103_13360 SL103_23275
SXI : SXIM_31500 SXIM_48060
STRC : AA958_01970 AA958_29905 AA958_32250
SCX : AS200_02580 AS200_04770 AS200_08945 AS200_10420 AS200_24165 AS200_38445
SRW : TUE45_01426(agaA_1) TUE45_06393(agaA_2) TUE45_06955(agaA_3) TUE45_07314(rafA)
SLE : sle_08850(sle_08850) sle_10440(sle_10440)
SCLF : BB341_02770 BB341_03155 BB341_14600 BB341_26335
SGS : AVL59_14270 AVL59_15200 AVL59_29960
STSI : A4E84_04785 A4E84_14775 A4E84_34815
SGM : GCM10017557_01410 GCM10017557_10140 GCM10017557_11310(galA) GCM10017557_79790
SCAD : DN051_08465 DN051_35495 DN051_37455
SLS : SLINC_7210 SLINC_7591 SLINC_8276 SLINC_8280
STRD : NI25_01255 NI25_34100
SNW : BBN63_02065 BBN63_04760 BBN63_04780
SAUO : BV401_00375 BV401_07785 BV401_33195 BV401_33215 BV401_37460 BV401_37770
SSIA : A7J05_05065 A7J05_30895 A7J05_33035
SPUN : BFF78_05130 BFF78_06375 BFF78_37940
SGV : B1H19_08025 B1H19_08630
SALF : SMD44_00426(melA) SMD44_01015 SMD44_07328
SLX : SLAV_02510(rafA1) SLAV_02535(rafA2) SLAV_06795(agaA) SLAV_36895(rafA3)
STRO : STRMOE7_06640 STRMOE7_07355 STRMOE7_32220
SNZ : DC008_02235 DC008_02255
SGE : DWG14_00695(agaA_1) DWG14_01392(agaSK_1) DWG14_01653(chiA2_3) DWG14_05307(galA) DWG14_05816(agaSK_2) DWG14_07374(agaA_2) DWG14_08149(agaSK_3) DWG14_08405
SLK : SLUN_20660 SLUN_34215
SDX : C4B68_04605 C4B68_28680 C4B68_38140
SGD : ELQ87_01100 ELQ87_02975 ELQ87_06815 ELQ87_37940
SCYA : EJ357_05190 EJ357_22150 EJ357_40610 EJ357_42205 EJ357_42260 EJ357_45630
SAST : CD934_03220 CD934_04935
SNQ : CP978_02190 CP978_02210 CP978_29000
SKA : CP970_04340 CP970_38530
SHAW : CEB94_05280 CEB94_15370 CEB94_35640
SGAL : CP966_34455 CP966_34875
SVR : CP971_28185 CP971_31965 CP971_31970 CP971_33055
SPAD : DVK44_01020 DVK44_30535 DVK44_35110 DVK44_35420
SFY : GFH48_06830 GFH48_06850 GFH48_35980 GFH48_37700
SAQU : EJC51_02755 EJC51_03435 EJC51_03620 EJC51_07650 EJC51_24000 EJC51_39535 EJC51_44880
SSEO : D0Z67_02265 D0Z67_26430
SRIM : CP984_33545 CP984_34705 CP984_35235
SPHV : F9278_05255 F9278_16940 F9278_40850
SCHA : CP983_00890 CP983_02185 CP983_02490 CP983_06370 CP983_22295 CP983_38450
SGX : H4W23_02385 H4W23_37000 H4W23_37280 H4W23_37340
SCOE : CP976_00460 CP976_00700 CP976_00715 CP976_04765 CP976_16325 CP976_37165
SSPB : CP982_34240 CP982_37845
SPLA : CP981_05250 CP981_05960 CP981_29825 CP981_32775
SBRO : GQF42_07030 GQF42_37680 GQF42_41775 GQF42_43745
SATA : C5746_00320 C5746_05560 C5746_35900 C5746_42090 C5746_42915
SHUN : DWB77_01396(agaSK) DWB77_06620(agaA_1) DWB77_06703(agaA_2)
SCYG : S1361_05560(agaA) S1361_33340(rafA)
SFEU : IM697_02045 IM697_18670 IM697_19930 IM697_19965 IM697_21170 IM697_28415
SLIA : HA039_00585 HA039_04720 HA039_15810
SDW : K7C20_04635 K7C20_05585 K7C20_28635 K7C20_31425
SAUH : SU9_003910 SU9_004555 SU9_027345
SROI : IAG44_04305 IAG44_06155 IAG44_41285 IAG44_41835
SHK : J2N69_04065 J2N69_32890
SANU : K7396_08445 K7396_30075 K7396_30825
SACT : DMT42_01310 DMT42_04430 DMT42_35850
SMAO : CAG99_01295 CAG99_01300 CAG99_15475
SINE : KI385_07730 KI385_08740 KI385_34255 KI385_37250
SLF : JEQ17_02730 JEQ17_06685 JEQ17_08650 JEQ17_42215 JEQ17_46475 JEQ17_46495
SDEC : L3078_32815 L3078_39075
SAOV : G3H79_00785 G3H79_04550 G3H79_33965
SDUR : M4V62_07565 M4V62_36785 M4V62_38290 M4V62_41160
SAKB : K1J60_05265 K1J60_09090 K1J60_27885
STAA : LDH80_03510 LDH80_07515
SNIG : HEK616_21750 HEK616_23140
SSPN : LXH13_01585 LXH13_04220 LXH13_05340 LXH13_05360 LXH13_35885
SXT : KPP03845_106234(agaA_2)
SLON : LGI35_08600 LGI35_39415 LGI35_43315
SDRZ : NEH16_02865 NEH16_28545
STUD : STRTU_006114 STRTU_006244
STEE : F3L20_19720 F3L20_19740
SANT : QR300_01690 QR300_02585 QR300_07405 QR300_09410 QR300_36680
SARG : HKX69_01310 HKX69_01330 HKX69_04470 HKX69_30855
SJN : RI060_03395 RI060_05700 RI060_38570
SCYN : N8I84_05770 N8I84_20015 N8I84_33015 N8I84_33900 N8I84_34310 N8I84_34370 N8I84_34645 N8I84_38950
SKG : KJK29_04140 KJK29_34115 KJK29_35630
SCOA : QU709_01525 QU709_04575 QU709_45295 QU709_45305
SLAI : P8A22_00745 P8A22_00830 P8A22_01995 P8A22_35085 P8A22_37395
SFP : QUY26_05685 QUY26_36045
SINN : ABB07_04885 ABB07_33435
KBU : Q4V64_38115 Q4V64_47165 Q4V64_48925
SARM : DVA86_20185 DVA86_21705
SPRF : K4G22_07490 K4G22_11680 K4G22_21480
SCT : SCAT_5636(agaA) SCAT_p0443(melA)
SCY : SCATT_56350 SCATT_p12960
KSK : KSE_06230 KSE_08070 KSE_67070
KAU : B6264_22885 B6264_24945 B6264_26150
KIT : CFP65_0498 CFP65_0582 CFP65_0928 CFP65_1274 CFP65_1591 CFP65_2272 CFP65_5772 CFP65_5856 CFP65_6723 CFP65_7188 CFP65_7210 CFP65_7374 CFP65_7398
STRI : C7M71_004370 C7M71_004910
STRH : GXP74_18665 GXP74_18685
ABRY : NYE86_14880 NYE86_19010 NYE86_19875 NYE86_20585 NYE86_22860 NYE86_23635
AGLA : OIE69_05830 OIE69_05930 OIE69_11085 OIE69_40610
PSIC : J4E96_08560 J4E96_13710
LEIF : HF024_02035(melA) HF024_03470 HF024_03475
LSE : F1C12_08940 F1C12_10065 F1C12_10800 F1C12_10810 F1C12_12310 F1C12_15510
LEIZ : IT072_01930 IT072_17730(melA) IT072_20410
CCAP : AES38_01010 AES38_01225 AES38_15050
CZH : H9X71_01080 H9X71_01090 H9X71_01335
MTS : MTES_1350 MTES_2021 MTES_2077 MTES_2872(aga)
MIM : AKG07_01895 AKG07_01900 AKG07_11850 AKG07_11955 AKG07_12350
MPAL : BO218_15890 BO218_15895
MICR : BMW26_06470 BMW26_06475 BMW26_16670 BMW26_17060
MAUR : BOH66_03095 BOH66_04650
MOO : BWL13_00191(rafA) BWL13_00439(agaSK) BWL13_01450(agaA)
MLZ : F6J85_06280 F6J85_12395
MRN : K8F61_10710 K8F61_11035
MICS : C1N74_11205 C1N74_11210
MBIN : LXM64_13040 LXM64_13955
MTEC : OAU46_11645 OAU46_15230
MARK : QUC20_11115 QUC20_11120
MRG : SM116_05630 SM116_05650 SM116_06560
MINV : T9R20_01775 T9R20_04025 T9R20_14530
MPLN : VC184_01220 VC184_10880 VC184_10885
RHOK : RKAS3_03980(galA_1) RKAS3_08780(galA_2)
RRY : C1O28_11695 C1O28_11705
RFS : C1I64_13845 C1I64_13995 C1I64_14005 C1I64_14925 C1I64_16785 C1I64_18165
RTE : GSU10_13650 GSU10_14830
ROE : Q0F99_10255 Q0F99_10270 Q0F99_10775
CUM : NI26_01005 NI26_01030 NI26_07535 NI26_11735
CUB : BJK06_01880 BJK06_13085 BJK06_14925 BJK06_14955
CUG : C1N91_00820 C1N91_00845 C1N91_07395 C1N91_11920
CCIT : QPK07_06020 QPK07_06055 QPK07_12115
CUY : I8920_11615 I8920_12260(melA) I8920_12270
FRP : AX769_03930 AX769_03945 AX769_11700
FSB : GCM10025867_00460 GCM10025867_20200(galA) GCM10025867_45190
AGY : ATC03_03505 ATC03_04675 ATC03_14775 ATC03_16040
AMAU : DSM26151_02800(palH)
ALAV : MTO99_12715 MTO99_17955
CART : PA27867_2680 PA27867_3367 PA27867_3385
CRY : B7495_01920 B7495_12980
MALK : MalAC0309_0323 MalAC0309_1515
MHUM : NNL39_04425 NNL39_11545
SALA : ESZ53_07765 ESZ53_10955
HUW : FPZ11_06865 FPZ11_12020 FPZ11_17180
LYD : D7I47_04770 D7I47_09005
PLAP : EAO79_01140 EAO79_03960 EAO79_04395 EAO79_05790 EAO79_13255 EAO79_13265
PFLU : BWO91_00210 BWO91_00705 BWO91_09620 BWO91_16820
HEA : HL652_14775(melA) HL652_14805(melA)
GLN : F1C58_00290 F1C58_11590
AGX : AGREI_1117 AGREI_1980(rafA)
ARE : AL755_09715 AL755_18495
ARW : MB46_00265 MB46_00720 MB46_13180
ARN : CGK93_02310 CGK93_18895
ACIT : HPK19_08035 HPK19_08105
AAGI : NCTC2676_1_01981(rafA)
APOA : J0916_04925 J0916_08855 J0916_13055
GAR : AOZ07_17505 AOZ07_17610
BCV : Bcav_0053 Bcav_0130 Bcav_0943 Bcav_2816
BFA : Bfae_05010 Bfae_07480 Bfae_29570
BRX : BH708_04700 BH708_17570
BGG : CFK41_01555 CFK41_13195 CFK41_14765
BSAU : DWV08_01045 DWV08_12340 DWV08_16480
BRR : C1N80_04220 C1N80_15095
BRHA : NLU66_00560 NLU66_04795 NLU66_16840
BHH : Bra3105_11765(melA) Bra3105_17320(melA)
LMOI : VV02_12205 VV02_19070 VV02_25770
XCE : Xcel_0260 Xcel_2118 Xcel_2915
IVA : Isova_0802 Isova_2425
IDO : I598_1451(agaA) I598_3475(rafA)
SKE : Sked_01850 Sked_01910 Sked_02790 Sked_31550 Sked_32900
CFL : Cfla_0456 Cfla_1094 Cfla_2742
CFI : Celf_0438 Celf_2718 Celf_2769 Celf_3623
CGA : Celgi_2554 Celgi_2570
CELZ : E5225_03940 E5225_12540 E5225_17005
CEJ : GC089_07185 GC089_08495
CELH : GXP71_02050 GXP71_02330 GXP71_10255 GXP71_18655
CIRA : LFM56_12765 LFM56_16325
CPAL : F1D97_01705 F1D97_16860
CDON : KKR89_02105 KKR89_05390
CWAN : KG103_02365 KG103_05740
CFEN : KG102_04110 KG102_09755 KG102_17995
CWN : NP075_02425 NP075_05610 NP075_08635
CCHE : NP064_05085 NP064_11345
CXIE : NP048_02565 NP048_05625
PSEK : GCM125_14820(galA) GCM125_15670
GEZ : FE251_04840 FE251_06365
HALT : IM660_11755(melA) IM660_12630 IM660_13020
RHAL : LQF10_06345 LQF10_06775 LQF10_07660(melA)
RSUA : LQF12_09970(melA) LQF12_10850 LQF12_11180
DSS : GCM25873_08010(galA)
ARUB : J5A65_03005 J5A65_08235 J5A65_08835
PBO : PACID_03120 PACID_06900 PACID_07300 PACID_32100
AACI : ASQ49_03525 ASQ49_06135
ACIJ : JS278_02992(rafA_1) JS278_02998(rafA_2)
MPH : MLP_15110 MLP_15200 MLP_27720 MLP_33590(aga) MLP_45180
MIK : FOE78_00295 FOE78_10085
MICG : GJV80_02025(melA) GJV80_02130 GJV80_07950 GJV80_19820
TEZ : BKM78_04855 BKM78_09895 BKM78_09915 BKM78_14165
TLA : TLA_TLA_00983(agaSK) TLA_TLA_01996(rafA_1) TLA_TLA_02000(galA) TLA_TLA_02852(rafA_2)
PRV : G7070_10525 G7070_16950
BRT : J4N02_06695 J4N02_08425
PSOI : PCC79_06770 PCC79_13610 PCC79_13650 PCC79_14755
NPS : KRR39_01700 KRR39_01765(melA)
NRN : SHK17_12335 SHK17_16495
KFL : Kfla_1515 Kfla_1749 Kfla_1996 Kfla_4308
KQI : F1D05_00840 F1D05_02180 F1D05_27670 F1D05_28455
KSL : OG809_01160 OG809_01165 OG809_03780 OG809_19505(melA) OG809_20540 OG809_27020 OG809_36015 OG809_38360
KRI : OX958_27620 OX958_28450 OX958_28750
ACTQ : OG417_12585 OG417_13590 OG417_16705 OG417_23740 OG417_33995 OG417_38485 OG417_47830
NOA : BKM31_03925 BKM31_03950 BKM31_06585 BKM31_10550 BKM31_16320 BKM31_16330 BKM31_19575 BKM31_20245 BKM31_29995 BKM31_30595 BKM31_49250 BKM31_56075
NOW : GBF35_03640 GBF35_28845 GBF35_29035 GBF35_30970 GBF35_32005 GBF35_34275 GBF35_34300 GBF35_48940
NGN : LCN96_14590 LCN96_19470(melA) LCN96_27375 LCN96_27385 LCN96_32420 LCN96_35025 LCN96_44890 LCN96_52240
NFS : OIE67_13435 OIE67_18265 OIE67_18275 OIE67_27165 OIE67_30640 OIE67_52065
MHAI : OHB01_07485 OHB01_27895 OHB01_32320
NAKA : H7F38_02270 H7F38_14120 H7F38_14220
SEN : SACE_3139(agaB2) SACE_7065(agaB2)
AMD : AMED_0853(galA) AMED_1747(galA) AMED_1851(galA) AMED_1923(galA) AMED_3645(galA) AMED_3750(galA) AMED_3913(galA) AMED_5042(galA) AMED_6804(galA) AMED_7386(galA) AMED_8989(galA)
AMN : RAM_04355 RAM_08870 RAM_09395 RAM_09755 RAM_18540 RAM_19095 RAM_19935 RAM_25670 RAM_34915 RAM_37970 RAM_46110
AMM : AMES_0851(galA) AMES_1733(galA) AMES_1837(galA) AMES_1907(galA) AMES_3604(galA) AMES_3705(galA) AMES_3867(galA) AMES_4982(galA) AMES_6702(galA) AMES_7275(galA) AMES_8853(galA)
AMZ : B737_0852(galA) B737_1734(galA) B737_1838(galA) B737_1908(galA) B737_3604(galA) B737_3705(galA) B737_3867(galA) B737_4982(galA) B737_6702(galA) B737_7275(galA) B737_8854(galA)
AOI : AORI_3230(galA) AORI_3670(galA)
AJA : AJAP_05700 AJAP_20810(galA) AJAP_23110
AMYC : CU254_08365 CU254_16330 CU254_25995
AMYB : BKN51_08560 BKN51_10825 BKN51_26690
AAB : A4R43_12120 A4R43_15645
AMYY : YIM_14815(agaA1) YIM_15990(agaA2) YIM_26800(agaA3) YIM_28715(agaA4) YIM_43295(agaA5)
AORI : SD37_07195 SD37_18845 SD37_20615 SD37_31500 SD37_36940
AACD : LWP59_07805 LWP59_10525
APRT : MUY14_02855 MUY14_12335 MUY14_21230 MUY14_26875 MUY14_27225 MUY14_27710 MUY14_38260 MUY14_45055 MUY14_45060
ARHD : VSH64_28705 VSH64_35310 VSH64_44250 VSH64_47185
AMOG : QRX60_01075 QRX60_01450 QRX60_01950 QRX60_13115 QRX60_31695 QRX60_39270 QRX60_39465
APRE : CNX65_05250 CNX65_08320 CNX65_15740 CNX65_18980 CNX65_30440
AMI : Amir_1044 Amir_1665 Amir_3106 Amir_3796 Amir_6181
SSYI : EKG83_16640 EKG83_21835 EKG83_29065 EKG83_32470
SACX : IOD16_22280 IOD16_22300 IOD16_31255
KAL : KALB_1281 KALB_1544 KALB_2845 KALB_3523 KALB_528 KALB_6161 KALB_6361 KALB_6880
KUT : JJ691_03890 JJ691_102970 JJ691_10790(galA_1) JJ691_27920 JJ691_32790(galA_2) JJ691_38700(galA_3) JJ691_39090 JJ691_50500(galA_4) JJ691_51280 JJ691_62080(galA_5)
KPHY : AOZ06_05440 AOZ06_13550 AOZ06_14370 AOZ06_39875
LED : BBK82_24450 BBK82_39615 BBK82_42220 BBK82_43095
AHM : TL08_15295 TL08_17380
ACTI : UA75_19000 UA75_21125
ACAD : UA74_18510 UA74_20630
AHG : AHOG_16380(rafA) AHOG_18410(agaA)
CROS : N8J89_05855 N8J89_09875 N8J89_12355 N8J89_15070 N8J89_17395
UME : RM788_03355 RM788_07670 RM788_08355 RM788_49330
MAU : Micau_1711 Micau_3680
MICB : MicB006_2780 MicB006_4702
MTUA : CSH63_06735 CSH63_15655
MICH : FJK98_08935 FJK98_09210 FJK98_14545 FJK98_14785 FJK98_14810
MTEM : GCE86_02860 GCE86_14545
MCAB : HXZ27_03205 HXZ27_09880 HXZ27_12960
MSAG : GCM10017556_01760(galA)
MENO : Jiend_58160(galA) Jiend_59440
MCHL : PVK74_08835 PVK74_29805
MFEU : H1D33_15465 H1D33_24235
MPRN : Q3V37_04140 Q3V37_19670 Q3V37_29515
ASE : ACPL_3009 ACPL_6037 ACPL_6723(rafA)
AMS : AMIS_27960 AMIS_28310 AMIS_29720 AMIS_29900 AMIS_64830
ACTN : L083_2615 L083_2951 L083_3265 L083_4155(rafA) L083_4539
AFS : AFR_14945 AFR_17105 AFR_18415 AFR_22550 AFR_29165
ACTS : ACWT_2537 ACWT_5905 ACWT_6593
AIH : Aiant_25250(galA_1) Aiant_73560(galA_2) Aiant_85540(galA_3)
ACTR : Asp14428_09220 Asp14428_14700(galA)
ACTL : L3i22_029980(galA) L3i22_041010(galA) L3i22_049680(galA) L3i22_049950(galA) L3i22_070600 L3i22_073040 L3i22_090210(galA)
ASIC : Q0Z83_015800 Q0Z83_027790 Q0Z83_028750 Q0Z83_049380 Q0Z83_051570 Q0Z83_105050
AOU : ACTOB_002758 ACTOB_004236 ACTOB_007013
PLAB : C6361_01335 C6361_22580 C6361_22695 C6361_28835 C6361_28840
PLAT : C6W10_17535 C6W10_18080 C6W10_18085 C6W10_31525
PFLA : Pflav_034320(galA) Pflav_079160
PSUU : Psuf_000080 Psuf_075840(galA_1) Psuf_078950(galA_2) Psuf_080560
ATL : Athai_12610 Athai_50120 Athai_56330 Athai_61920(galA)
ASER : Asera_13480(galA) Asera_18540 Asera_24450 Asera_35270 Asera_49490 Asera_61100
CATI : CS0771_42580 CS0771_59410(galA_1) CS0771_63430 CS0771_66610(galA_2)
DVC : Dvina_10475 Dvina_14990 Dvina_19195 Dvina_23225 Dvina_24360 Dvina_24430 Dvina_37955 Dvina_45550
DFU : Dfulv_02110 Dfulv_19795
DAUR : Daura_18655 Daura_32165 Daura_37430 Daura_43860
DMAT : Dmats_10045 Dmats_14450 Dmats_18765 Dmats_19985 Dmats_20055 Dmats_32975 Dmats_39915
DACT : KZZ52_36160 KZZ52_36300 KZZ52_46080 KZZ52_51955 KZZ52_57415
NHY : JQS43_11700 JQS43_22365
CCAZ : COUCH_13760 COUCH_23315 COUCH_25170
CAI : Caci_2552 Caci_4439 Caci_4641 Caci_4693 Caci_4912 Caci_5007 Caci_6252 Caci_6873 Caci_6874 Caci_7223 Caci_7918
GLY : K3N28_00085 K3N28_00155 K3N28_07260 K3N28_09915
TPYO : X956_00995 X956_10185
TPEC : HLG82_09005 HLG82_09485
ARD : AXF14_00895 AXF14_12700
ACTC : CHIBA101_1596 CHIBA101_1998
ANE : ATCC27039_05870(galA) ATCC27039_23740
AHW : NCTC11636_00064(rafA) NCTC11636_01732
AIR : NCTC12972_01877(rafA)
ASLA : NCTC11923_00522(rafA_1) NCTC11923_01103(rafA_2)
AVC : NCTC10951_00363(rafA) NCTC10951_02313
AMAM : HPC72_01845 HPC72_06720
ACAP : MANAM107_11500 MANAM107_20180(galA)
AUO : R3I39_05615 R3I39_06910
ALIL : D5R93_08630 D5R93_12860
GHM : CJ187_004025 CJ187_006200
BLX : GS08_09910 GS08_10375
BAD : BAD_1525 BAD_1528 BAD_1576(aga)
BADL : BADO_0439 BADO_1484 BADO_1682
BADO : BBMN23_1680 BBMN23_1748
BLA : BLA_0881 BLA_1524(aga)
BLC : Balac_1537 Balac_1601
BLT : Balat_1537 Balat_1601
BNM : BALAC2494_01144 BALAC2494_01213
BANI : BL12_07655 BL12_07960
BANL : BLAC_07660 BLAC_07950
BNI : BANAN_07390 BANAN_07690
BANM : EN10_07770 EN10_08080
BDE : BDP_0134 BDP_0227 BDP_0487 BDP_2092 BDP_2102(aga) BDP_2229(aga)
BDN : BBDE_0124 BBDE_0215 BBDE_0465 BBDE_1968 BBDE_1976 BBDE_2095
BBV : HMPREF9228_0308 HMPREF9228_1959
BBRE : B12L_0243 B12L_1807
BBRV : B689b_0272 B689b_1893
BBRJ : B7017_0265 B7017_2073
BBRC : B7019_0275 B7019_2060
BBRN : B2258_0272 B2258_1876
BBRS : BS27_0297 BS27_1854(aga)
BBRD : BBBR_0247 BBBR_1868
BAST : BAST_0057 BAST_0058
BCOR : BCOR_0125 BCOR_0138
BII : BINDI_0028 BINDI_0029
BKA : AH68_09535 AH68_09925
BKS : BBKW_1788 BBKW_1856 BBKW_1864
BCAT : BBCT_0489 BBCT_1564 BBCT_1646
BPSP : AH67_00735 AH67_08185 AH67_08535
BANG : BBAG_0017 BBAG_0702
BPSC : BBPC_1670 BBPC_1797 BBPC_1812
BSCA : BBSC_0162 BBSC_0165 BBSC_0906 BBSC_1607 BBSC_1615 BBSC_1618 BBSC_1866 BBSC_2046 BBSC_2430 BBSC_2522
BACT : AB656_05960 AB656_06235 AB656_06395
BCHO : BcFMB_01830 BcFMB_02250 BcFMB_03150
BGX : ESN35_00135 ESN35_00530 ESN35_10075
BLEM : BL8807_01475 BL8807_03460 BL8807_03715
BEU : BE0216_07905 BE0216_09860 BE0216_09920
BFJ : B19861_17590 B19861_18140
JTL : M6D93_09790(melA) M6D93_15390 M6D93_18315
RUB : GBA63_13180(melA) GBA63_13185(melA)
BALA : DSM104299_04095(melA)
CWO : Cwoe_3958 Cwoe_4885 Cwoe_5795
IAM : HC251_07120 HC251_09235
RRS : RoseRS_1356 RoseRS_1360 RoseRS_2891
RCA : Rcas_1884 Rcas_3563 Rcas_3567
ATM : ANT_04010 ANT_08810 ANT_08820(melA) ANT_25810
CAP : CLDAP_02770 CLDAP_02780 CLDAP_08940 CLDAP_12840 CLDAP_12850(melA)
KBS : EPA93_01090(melA) EPA93_06135(melA)
TTR : Tter_0137 Tter_0216 Tter_2324 Tter_2439 Tter_2514 Tter_2515
PMET : G4Y79_03025 G4Y79_03095(melA) G4Y79_09520 G4Y79_18455
DGO : DGo_CA1339 DGo_CA1340(agaL)
DPD : Deipe_0042 Deipe_0778 Deipe_1344
DEIN : DAAJ005_03965 DAAJ005_16245
DPH : EHF33_14550 EHF33_16575 EHF33_17985
FGI : OP10G_0046 OP10G_0375 OP10G_1040 OP10G_2050
CCOT : CCAX7_008810 CCAX7_20050 CCAX7_24660 CCAX7_58730
OTE : Oter_1363 Oter_2301 Oter_3220
OBG : Verru16b_02656(agaA)
VBH : CMV30_05145 CMV30_15700 CMV30_16545 CMV30_17205
OLE : K0B96_01225 K0B96_05695 K0B96_14815
SLOM : PXH66_03755 PXH66_11845
ELUT : CKA38_02950 CKA38_07390 CKA38_08770 CKA38_14215
AAQW : K1X11_013185 K1X11_022035
TMV : OH491_03270 OH491_05930 OH491_14860 OH491_15190 OH491_24090 OH491_25260
OBT : OPIT5_17570 OPIT5_17825 OPIT5_18930 OPIT5_30620 OPIT5_30755
CALG : SH580_01910 SH580_16995
RUFI : K0V07_02600 K0V07_07310 K0V07_16155
ABIW : Abiwalacus_09310 Abiwalacus_09590
AMAK : J5W79_03940 J5W79_05640 J5W79_05805
LUO : HHL09_07915 HHL09_13905
LAMB : KBB96_04420 KBB96_11095 KBB96_16030 KBB96_16740
LUI : llg_14520 llg_25480 llg_27860
VEG : SAMN05444156_1716 SAMN05444156_2991
VBA : IMCC26134_04990 IMCC26134_05140 IMCC26134_08335 IMCC26134_08915
VBS : EGM51_03195 EGM51_06215
LPAV : PLANPX_1437 PLANPX_2481 PLANPX_3310 PLANPX_3340 PLANPX_3342 PLANPX_3347
AMUC : Pan181_51470(agaA_2)
PND : Pla175_00120(agaA_1) Pla175_44510(agaA_2)
PHM : PSMK_02000 PSMK_08660
PBP : STSP1_01036(agaA) STSP1_02422(rafA)
ALUS : STSP2_00372(agaA_1) STSP2_01237(rafA_2) STSP2_02148(agaA_2) STSP2_02617(agaA_3)
VBC : C5Q97_00940 C5Q97_01230 C5Q97_08055 C5Q97_08310 C5Q97_11160 C5Q97_15270 C5Q97_18425 C5Q97_19125 C5Q97_21170 C5Q97_21210
TRC : DYE49_08020 DYE49_12040
SSM : Spirs_0159 Spirs_0837 Spirs_1236
SLR : L21SP2_2760 L21SP2_2809
OCK : EXM22_06270 EXM22_13895
SBU : SpiBuddy_0436 SpiBuddy_0585
SASS : MUG09_02365 MUG09_03300
SCC : Spico_0769 Spico_1068
TAZ : TREAZ_0745 TREAZ_0999 TREAZ_2287 TREAZ_2577
SCD : Spica_0299 Spica_2220 Spica_2490
BHC : JFL75_19970 JFL75_20285
ABA : Acid345_0664 Acid345_0944 Acid345_1677 Acid345_3249
ACA : ACP_0049 ACP_0066(agaN) ACP_0856 ACP_1583
ACM : AciX9_1740 AciX9_1741 AciX9_3820 AciX9_4314
GMA : AciX8_0850 AciX8_0890 AciX8_0954 AciX8_2438 AciX8_2696 AciX8_2697 AciX8_2904 AciX8_3428 AciX8_3580 AciX8_4018 AciX8_4774
GRW : FTO74_09600 FTO74_09605 FTO74_09785
GRB : GOB94_05330 GOB94_09495 GOB94_11775
TSA : AciPR4_1545 AciPR4_1700 AciPR4_2725 AciPR4_4094
TRS : Terro_1771 Terro_2888 Terro_4303
TALB : FTW19_05995 FTW19_08005 FTW19_09870 FTW19_09920 FTW19_17040 FTW19_17045 FTW19_20975 FTW19_21320 FTW19_23600
ABAS : ACPOL_0350 ACPOL_0404 ACPOL_1588 ACPOL_1747 ACPOL_1808 ACPOL_2237 ACPOL_2606 ACPOL_4612 ACPOL_4675 ACPOL_6713
EDA : GWR55_06070 GWR55_12770 GWR55_13015
EDG : H7846_05585 H7846_13670
ADIN : H7849_08025 H7849_08750 H7849_08775 H7849_25290 H7849_26260
ORP : MOP44_07115 MOP44_17155
TGI : RBB81_02540 RBB81_05825 RBB81_08390 RBB81_08415 RBB81_13955 RBB81_14765
TEMP : RBB75_00365 RBB75_08610 RBB75_10340
TPSC : RBB77_01485 RBB77_03750 RBB77_04505 RBB77_04525 RBB77_22975
SUS : Acid_0705 Acid_0933 Acid_1472 Acid_1588 Acid_2820 Acid_3941 Acid_7394
PFER : IRI77_04670 IRI77_05970 IRI77_09910 IRI77_11485 IRI77_32850 IRI77_35480(melA) IRI77_37345
FMO : C4N19_07040 C4N19_09615
CSOM : MKD34_09385 MKD34_13065
GBA : J421_0655 J421_4530 J421_5118 J421_5651
BTH : BT_2851 BT_3065 BT_3131 BT_3133 BT_3592
BTHO : Btheta7330_00053(agaA_1) Btheta7330_04693(agaA_4) Btheta7330_05148 Btheta7330_05150(rafA)
BFR : BF0287 BF0550 BF1485 BF4391
BFS : BF9343_0230 BF9343_0475 BF9343_1347 BF9343_4081
BFG : BF638R_0288(aga) BF638R_0546(aga) BF638R_1499 BF638R_4272
BFB : VU15_00770 VU15_01925 VU15_06225 VU15_20345
BXY : BXY_19030 BXY_19750 BXY_20040 BXY_26070 BXY_26080 BXY_27370
BOA : Bovatus_02926(rafA_1) Bovatus_02938(rafA_2) Bovatus_03161(rafA_3) Bovatus_03546(rafA_4)
BCEL : BcellWH2_00425(rafA_1) BcellWH2_00729(agaA_1) BcellWH2_01206(rafA_2) BcellWH2_01331(rafA_3) BcellWH2_02847(agaA_2) BcellWH2_03485(rafA_4)
BCAC : CGC64_03505 CGC64_04690
BCAE : A4V03_00060 A4V03_14955 A4V03_18990 A4V03_18995 A4V03_19440
BIS : DXK01_002230 DXK01_005025 DXK01_018930 DXK01_020555 DXK01_021300
BUN : Bun01g_01040 Bun01g_02210(galA) Bun01g_23620
BFE : K6V30_03790 K6V30_04065 K6V30_04375 K6V30_04385
BNO : K8P02_03325 K8P02_06630 K8P02_13445 K8P02_16650 K8P02_16675 K8P02_17650 K8P02_17935
BHUM : JXR92_000490 JXR92_009890 JXR92_009905 JXR92_010740 JXR92_011590
BFC : BacF7301_03215 BacF7301_06470
BFIN : BFINE_11860 BFINE_14760
BSA : Bacsa_1703 Bacsa_2837
BDO : EL88_22985 EL88_23020
BDH : GV66_03470 GV66_03505
MICA : P0L94_00070 P0L94_18395
PPN : Palpr_1626 Palpr_1630 Palpr_1815
PARC : CI960_03555 CI960_12805
PGOL : K6V26_09745 K6V26_21820 K6V26_23250 K6V26_23255 K6V26_23270
PJO : LK449_07505 LK449_10590
PFAI : NXY11_08735 NXY11_20700 NXY11_22315 NXY11_22320 NXY11_22335
PARY : A4V02_01335 A4V02_08955 A4V02_09085
DUN : FDZ78_04235 FDZ78_09130
DUQ : E7745_00530 E7745_05425
BVS : BARVI_03220 BARVI_10620
COPR : Cop2CBH44_02760 Cop2CBH44_02770 Cop2CBH44_03100 Cop2CBH44_17720 Cop2CBH44_23370 Cop2CBH44_27150 Cop2CBH44_28530
CFAS : Cfast33896_13080 Cfast33896_13220 Cfast33896_26480
PDT : Prede_1307 Prede_1308 Prede_1617 Prede_1618 Prede_2024
PRO : HMPREF0669_00808 HMPREF0669_00811
PFUS : ADJ77_05440 ADJ77_07260
PMUF : J4864_03390 J4864_05980 J4864_09130
PVF : J5A55_00645 J5A55_08590
POC : NCTC13071_01506(agaA)
SHOI : KUA50_011580 KUA50_013985
PCLR : PC1C4_04550 PC1C4_04560
ASH : AL1_00380 AL1_15850 AL1_16560 AL1_25580
ALD : GFH31_05310 GFH31_05315 GFH31_07755 GFH31_10330
AOK : A3BBH6_11460 A3BBH6_28710
ACOU : A5CBH24_02150 A5CBH24_13040
ADA : A5CPEGH6_11100 A5CPEGH6_12420 A5CPEGH6_15780
AIT : AI2BBH_01390 AI2BBH_03470 AI2BBH_23790
ASEN : NQ519_00345 NQ519_06400
DORI : FH5T_01990 FH5T_04355 FH5T_07615 FH5T_07695
DRC : G0Q07_02050 G0Q07_17670
MCOS : GM418_01450 GM418_01925 GM418_06700 GM418_09265 GM418_10765 GM418_14220 GM418_21595 GM418_23395
ANF : AQPE_0837 AQPE_1314 AQPE_2596 AQPE_4511
CPI : Cpin_3595 Cpin_5645 Cpin_5646 Cpin_5740
CHIT : FW415_12185 FW415_14130 FW415_17455 FW415_20630
CHIH : GWR21_05400 GWR21_08730 GWR21_08735 GWR21_09030
COY : HF329_11470 HF329_21160 HF329_21635 HF329_25290
CFIL : MYF79_07785 MYF79_19900 MYF79_20055 MYF79_20060
CHF : KTO58_08060 KTO58_08065
CHOR : MKQ68_02955 MKQ68_07010
CSAC : SIO70_20470 SIO70_26190
NKO : Niako_0680 Niako_1468 Niako_1475 Niako_3453 Niako_5379
NSO : NIASO_02885 NIASO_08890 NIASO_10935 NIASO_16065
NIA : A8C56_11815 A8C56_20520 A8C56_22030
NYN : U0035_01170 U0035_20410 U0035_20455
FGG : FSB75_00145 FSB75_00250 FSB75_00495 FSB75_06410 FSB75_09610 FSB75_10460
ARB : A9P82_02800 A9P82_02845 A9P82_02880 A9P82_02920 A9P82_04630
ARK : D6B99_01220 D6B99_05380 D6B99_07650 D6B99_09165 D6B99_11940
AGI : FSB73_00315 FSB73_04780 FSB73_07195 FSB73_07210 FSB73_08045 FSB73_09040 FSB73_10880 FSB73_12240 FSB73_12245 FSB73_16945 FSB73_21245 FSB73_22740
ATEE : K9M52_07710 K9M52_07720 K9M52_09480 K9M52_12140 K9M52_12145
FLN : FLA_2823 FLA_3977 FLA_3978 FLA_5529 FLA_6374
PSEG : D3H65_02765 D3H65_05815 D3H65_05845
PGO : FSB84_14645 FSB84_14675 FSB84_14685 FSB84_17725
FLC : KJS93_11550 KJS93_17895
HHY : Halhy_3134 Halhy_3136 Halhy_6804
PHE : Phep_0715 Phep_1706 Phep_1839 Phep_2885 Phep_2887
PEP : AQ505_11245 AQ505_15485 AQ505_15490 AQ505_15675 AQ505_17305
PSTY : BFS30_00195 BFS30_03740 BFS30_03745 BFS30_21025
PGS : CPT03_04810 CPT03_06730 CPT03_08245 CPT03_12235 CPT03_13470
PEK : FFJ24_005985 FFJ24_009210 FFJ24_009215 FFJ24_012720
PROE : H9L23_04975 H9L23_11920 H9L23_11925 H9L23_14700
PRK : H9N25_04335 H9N25_10185
PGJ : QG516_00305 QG516_04375
PSN : Pedsa_0496 Pedsa_2573 Pedsa_3616
SHG : Sph21_4066 Sph21_4263
SPHN : BV902_06600 BV902_22515 BV902_22535
SPDR : G6053_06450 G6053_06470
STHA : NCTC11429_02784(rafA)
SSIY : JVX97_08695 JVX97_08715
SCN : Solca_0509 Solca_1514 Solca_2503
SLAS : L2B55_03680 L2B55_05330 L2B55_06965
MUP : A0256_02480 A0256_07630 A0256_12980
MUC : MuYL_0177 MuYL_0984 MuYL_2048 MuYL_4390
MGOT : MgSA37_00434(rafA) MgSA37_00733(agaA_1) MgSA37_00734(agaA_2) MgSA37_02271 MgSA37_03489(agaA_3) MgSA37_03673(agaA_4)
MUH : HYN43_000770 HYN43_001855 HYN43_014620 HYN43_028770
MGIN : FRZ54_05520 FRZ54_10695 FRZ54_11635 FRZ54_16265
MGK : FSB76_04235 FSB76_17965 FSB76_21760 FSB76_26765 FSB76_28250 FSB76_28255
MRUB : DEO27_000645 DEO27_002540 DEO27_008135 DEO27_014800 DEO27_016500 DEO27_017290 DEO27_027575 DEO27_030485
MGOS : DIU38_001015 DIU38_014935 DIU38_017715 DIU38_019230 DIU38_032710
MMAB : HQ865_01955 HQ865_10845 HQ865_15370 HQ865_15690 HQ865_18730 HQ865_19475 HQ865_23745
MDJ : LLH06_11840 LLH06_13295
MJJ : PQO05_07315 PQO05_13105
MSAB : SNE25_05880 SNE25_10920 SNE25_15360 SNE25_23335 SNE25_23340 SNE25_26790
OLI : FKG96_17465 FKG96_18785
SBX : CA265_04030 CA265_05975 CA265_10275 CA265_10280 CA265_11135 CA265_24330
SPIK : EXU85_07165 EXU85_19145 EXU85_19155
SENF : GJR95_23735 GJR95_23745 GJR95_29365 GJR95_32090
EOL : Emtol_1007 Emtol_3305 Emtol_3307
EMZ : MB380_14635 MB380_14640
FAE : FAES_0887(galA1) FAES_2384(galA3) FAES_4801
HYZ : AXW84_10370 AXW84_10380
HYJ : FHG12_15580 FHG12_16655 FHG12_20625
HTB : MTX78_07395 MTX78_17985
HVL : MUN86_15580 MUN86_25740
RTI : DC20_05270 DC20_13720
FLM : MY04_4375 MY04_4445 MY04_5235
FYA : KMW28_21835 KMW28_24155 KMW28_24495
FBT : D770_13135 D770_20085 D770_23570
RCG : N7E81_07700 N7E81_07725 N7E81_08235 N7E81_12170
FAX : FUAX_10070 FUAX_41580 FUAX_51980
GRS : C7S20_04560 C7S20_18805
FJG : BB050_02616(agaA_1) BB050_03259(agaA_2) BB050_03399 BB050_03400 BB050_03402(rafA_1) BB050_03529(rafA_2)
FCM : BIW12_09755 BIW12_09780 BIW12_12165
FCR : HYN56_20785 HYN56_24355
FNK : E1750_16695 E1750_17270 E1750_17300 E1750_17555
FEN : J0383_16510 J0383_20580
FPB : NLJ00_00930 NLJ00_00940 NLJ00_00945 NLJ00_20765 NLJ00_24385
FBI : L0669_14710 L0669_14720 L0669_15300 L0669_17835 L0669_18740 L0669_23015
FHU : M0M44_18750 M0M44_18760 M0M44_18765 M0M44_19870 M0M44_23190
FPEC : Q1W71_03675 Q1W71_07770 Q1W71_10835
FGC : LNP27_14015 LNP27_14025
ZGA : ZOBELLIA_1055 ZOBELLIA_2089 ZOBELLIA_3523
POA : CW731_00370 CW731_00480
MARF : CJ739_2418 CJ739_722
FBE : FF125_04040 FF125_07045
HWS : RNZ46_12490 RNZ46_12515
ORH : Ornrh_0266 Ornrh_1411
ORI : Q785_01445 Q785_02870
EMN : M876_05325 M876_05355
EEN : BBD30_18535 BBD30_18570
ELB : VO54_01130(rafA) VO54_01136(agaA)
EMG : BBD33_14110 BBD33_14140
EGO : BBD34_16330 BBD34_16360
EGM : AYC65_10265 AYC65_10295
CGLE : NCTC11432_01200(agaA_1)
MINF : MESINF_0988 MESINF_1050(GalA)
ASAC : ATHSA_0378 ATHSA_1091 ATHSA_1658
CABY : Cabys_1671 Cabys_2804
DTH : DICTH_0117 DICTH_0275 DICTH_1562
HDS : HSR122_2563(celF) HSR122_2564
HRR : HZS55_01560(melA) HZS55_13740(melA)
HAAD : MW046_14335 MW046_18455(melA)
HTU : Htur_4484 Htur_4649 Htur_4675
HSAL : JMJ58_21215 JMJ58_21355
HXA : Halxa_0505 Halxa_3609
» show all Taxonomy Reference Authors Suzuki H, Li SC, Li YT.
Title Alpha-galactosidase from Mortierella vinacea. Crystallization and properties.
Journal J Biol Chem 245:781-6 (1970)
Reference Authors Vidershain GIa, Beier EM.
Title [Interrelation of alpha-D-fucosidase and alpha-D-galactosidase activities in man and animals]
Journal Dokl Akad Nauk SSSR 231:486-8 (1976)
Other DBs ExplorEnz - The Enzyme Database: 3.2.1.22
ExPASy - ENZYME nomenclature database: 3.2.1.22