KEGG の概要
1. ゲノムから生命システムへ
KEGG はゲノムや分子レベルの情報から細胞、個体、生物界といった生命システムがもつ高次の機能や有用性を理解するためのリソースです。生命システムのコンピュータモデルとして、遺伝子やタンパク質(ゲノム情報)と化合物など(ケミカル情報)の分子部品の情報を、分子間の配線図である相互作用・反応ネットワーク(システム情報)で統合した形で表現されています。さらに疾患・医薬品情報(ヘルス情報)もゆらいだ分子ネットワークとして統合されています。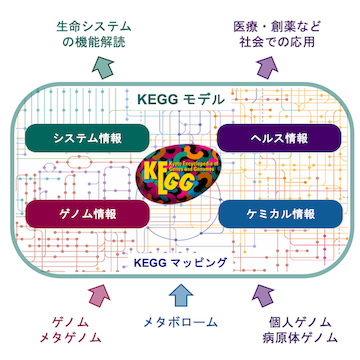
2. KEGG データベース
KEGG モデルは以下の 16のデータベースを統合したリソースとして実装されています。これらはシステム情報、ゲノム情報、ケミカル情報、ヘルス情報に大別され、さらに各ウェブページでは 5つのカラーコードで区別されています。| カテゴリ | データベース | 内容 | カラー |
| システム情報 | KEGG PATHWAY | KEGG パスウェイマップ | |
| KEGG BRITE | BRITE 機能階層・テーブル | ||
| KEGG MODULE | KEGG モジュールと反応モジュール | ||
| ゲノム情報 | KEGG ORTHOLOGY (KO) | 機能オーソログ | |
| KEGG GENES | 遺伝子・タンパク質 | ||
| KEGG GENOME | 全ゲノム配列既知の KEGG 生物種 | ||
| ケミカル情報 | KEGG COMPOUND | 代謝物その他の化学物質 | |
| KEGG GLYCAN | 糖鎖 | ||
| KEGG REACTION KEGG RCLASS |
生体内化学反応 反応クラス | ||
| KEGG ENZYME | 酵素 | ||
| ヘルス情報 | KEGG NETWORK | 疾患関連のネットワーク要素 | |
| KEGG VARIANT | ヒト遺伝子バリアント | ||
| KEGG DISEASE | ヒト疾患 (日本語) | ||
| KEGG DRUG KEGG DGROUP |
医薬品 (日本語) 医薬品グループ (日本語) |
これらのデータベースには生命システムのコンピュータ表現として様々なデータオブジェクトが含まれています。各データベースのエントリは KEGG オブジェクトと呼ばれ、データベースごとに定められたプリフィックスと5桁の数字からなる KEGG オブジェクト識別子がエントリ名となっています (KEGG Objects 参照)。
| リリース | データベース | オブジェクト識別子 | 注記 |
| 1995 | KEGG PATHWAY | map number | |
| KEGG GENES | locus_tag / GeneID | ||
| KEGG ENZYME | EC number | ||
| KEGG COMPOUND | C number | ||
| 1998 | KEGG REACTION | R number | |
| 2000 | KEGG GENOME | organism code / T number | |
| 2002 | KEGG ORTHOLOGY | K number | Ortholog IDs in 2000 |
| 2003 | KEGG GLYCAN | G number | |
| 2004 | KEGG RPAIR | RP number | Discontinued in 2016 |
| 2005 | KEGG BRITE | br number | |
| KEGG DRUG | D number | ||
| 2006 | KEGG MODULE | M number | |
| 2008 | KEGG DISEASE | H number | |
| 2010 | KEGG RCLASS | RC number | |
| KEGG EDRUG | E number | Renamed to ENVIRON | |
| 2011 | KEGG ENVIRON | E number | Discontinued in 2021 |
| 2014 | KEGG DGROUP | DG number | |
| 2017 | KEGG NETWORK | N number / nt number | |
| KEGG VARIANT | GeneID+variant number |
3. KEGG 分子ネットワーク
KEGG で最もユニークなデータベースは高次生命システム機能に関する知識を分子間相互作用・反応・関係ネットワークとして表現したシステム情報の部分です。ここでは文献等から集約した知識を以下の表現でコンピュータ化しています。- パスウェイマップ - KEGG PATHWAY (Pathway maps 参照)
- BRITE 階層・テーブル - KEGG BRITE (Brite hierarchies 参照)
- メンバーシップ (論理式) - KGG MODULE
- メンバーシップ (単純リスト) - KEGG DISEASE
このようなマッピングの概念は 1995年に KEGG プロジェクトが開始されたときに初めて導入され実用化されました。当初は EC 番号を用いてゲノムから代謝パスウェイ再構築を行っていましたが、その後代謝系以外の様々なパスウェイに対応するために、また EC 番号の不備を補うために、ortholog ID と呼ぶ識別子が導入されました。現在はこれをさらに見直し拡張した KO (KEGG Orthology) システムを用いてゲノムアノテーションと KEGG 分子ネットワークへのマッピングが行われています。
| 時期 | 識別子 | 知識ベース | 割当単位 |
| 1995-1999 | EC 番号 | 代謝パスウェイ | ドメイン |
| 2000-2002 | Ortholog ID | 代謝・制御パスウェイ | ドメイン |
| 2003- | KO | パスウェイおよび BRITE 機能階層 | 遺伝子 |
別の見方をすると、KO は分子ネットワーク上でのオーソログとして定義され、個々の遺伝子情報を一般化したものです。KEGG にはこれ以外にも同様の一般化が、特殊な分子ネットワークについて行われています。
| ネットワークタイプ | クラス | インスタンス |
| すべてのタイプ | KO (遺伝子オーソログ) | 遺伝子 in KEGG GENES |
| 生体内化学反応 | RC (反応クラス) | 反応 in KEGG REACTION |
| 医薬品相互作用 | DG (医薬品グループ) | 医薬品 in KEGG DRUG |
4. ネットワークバリアント
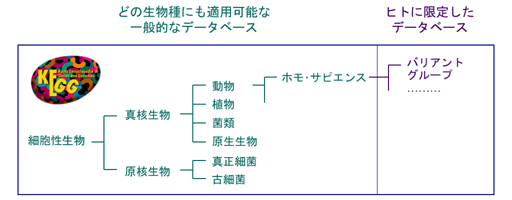
これまで KEGG データベースでは、遺伝子やゲノムについて生物種間での保存性と多様性に着目した開発を行ってきました。とくに KEGG パスウェイマップ、BRITE 機能階層、KEGG モジュールを、機能オーソログ (KO) をノードとしたネットワークとして開発することにより、KEGG パスウェイマッピングその他の手続きがどんな生物種にも適用できるようにしました。
参考文献
- Kanehisa, M.; Toward pathway engineering: a new database of genetic and molecular pathways. Science & Technology Japan, No. 59, pp. 34-38 (1996). [pdf]
- Kanehisa, M.; A database for post-genome analysis. Trends Genet. 13, 375-376 (1997). [pubmed] [doi]
- Ogata, H., Goto, S., Sato, K., Fujibuchi, W., Bono, H., and Kanehisa, M.; KEGG: Kyoto Encyclopedia of Genes and Genomes. Nucleic Acids Res. 27, 29-34 (1999). [pubmed] [doi]
- Kanehisa, M. and Goto, S.; KEGG: Kyoto Encyclopedia of Genes and Genomes. Nucleic Acids Res. 28, 27-30 (2000). [pubmed] [doi]
- Kanehisa, M., Goto, S., Kawashima, S., and Nakaya, A.; The KEGG databases at GenomeNet. Nucleic Acids Res. 30, 42-46 (2002). [pubmed] [doi]
- Kanehisa, M., Goto, S., Kawashima, S., Okuno, Y., and Hattori, M.; The KEGG resources for deciphering the genome. Nucleic Acids Res. 32, D277-D280 (2004). [pubmed] [doi]
- Kanehisa, M., Goto, S., Hattori, M., Aoki-Kinoshita, K.F., Itoh, M., Kawashima, S., Katayama, T., Araki, M., and Hirakawa, M.; From genomics to chemical genomics: new developments in KEGG. Nucleic Acids Res. 34, D354-357 (2006). [pubmed] [doi]
- Kanehisa, M., Araki, M., Goto, S., Hattori, M., Hirakawa, M., Itoh, M., Katayama, T., Kawashima, S., Okuda, S., Tokimatsu, T., and Yamanishi, Y.; KEGG for linking genomes to life and the environment. Nucleic Acids Res. 36, D480-D484 (2008). [pubmed] [doi]
- Kanehisa, M., Goto, S., Furumichi, M., Tanabe, M., and Hirakawa, M.; KEGG for representation and analysis of molecular networks involving diseases and drugs. Nucleic Acids Res. 38, D355-D360 (2010). [pubmed] [doi]
- Kanehisa, M., Goto, S., Sato, Y., Furumichi, M., and Tanabe, M.; KEGG for integration and interpretation of large-scale molecular datasets. Nucleic Acids Res. 40, D109-D114 (2012). [pubmed] [doi]
- Kanehisa, M., Goto, S., Sato, Y., Kawashima, M., Furumichi, M., and Tanabe, M.; Data, information, knowledge and principle: back to metabolism in KEGG. Nucleic Acids Res. 42, D199–D205 (2014). [pubmed] [doi]
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., and Tanabe, M.; KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44, D457-D462 (2016). [pubmed] [doi]
- Kanehisa, Furumichi, M., Tanabe, M., Sato, Y., and Morishima, K.; KEGG: new perspectives on genomes, pathways, diseases and drugs. Nucleic Acids Res. 45, D353-D361 (2017). [pubmed] [doi]
- Kanehisa, M., Sato, Y., Furumichi, M., Morishima, K., and Tanabe, M.; New approach for understanding genome variations in KEGG. Nucleic Acids Res. 47, D590-D595 (2019). [pubmed] [doi]
- Kanehisa, M; Toward understanding the origin and evolution of cellular organisms. Protein Sci. 28, 1947-1951 (2019). [pubmed] [doi]
- Kanehisa, M., Furumichi, M., Sato, Y., Ishiguro-Watanabe, M., and Tanabe, M.; KEGG: integrating viruses and cellular organisms. Nucleic Acids Res. 49, D545-D551 (2021). [pubmed] [doi]
- Kanehisa, M., Furumichi, M., Sato, Y., Kawashima, M. and Ishiguro-Watanabe, M.; KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023). [pubmed] [doi]
- Kanehisa, M., Furumichi, M., Sato, Y., Matsuura, Y. and Ishiguro-Watanabe, M.; KEGG: biological systems database as a model of the real world. Nucleic Acids Res. 53, D672-D677 (2025). [pubmed] [doi]
Last updated: January 7, 2025
